アルゴノート (タンパク質)
アルゴノート(英: Argonaute)タンパク質ファミリーは、RNA誘導サイレンシング複合体(RISC)の必須構成要素として、RNAサイレンシングプロセスにおいて中心的な役割を果たしている。RISCは、RNA干渉(RNAi)として知られる遺伝子サイレンシング現象を担っている。アルゴノートタンパク質は、マイクロRNA(miRNA)、低分子干渉RNA(siRNA)、Piwi結合RNA(piRNA)など、さまざまなクラスの低分子非コードRNAと結合する。低分子RNAは、配列相補性(塩基対形成)を通じてアルゴノートタンパク質を特定の標的へ誘導し、次にmRNAを切断または翻訳の抑制を引き起こす。
このタンパク質ファミリーの名前は、Bohmertらによって、シロイヌナズナ(Arabidopsis thaliana)のAGO1の突然変異による表現型が、遠洋性タコのアオイガイ(Argonauta argo)の外観に例えられたことに由来する[1]。
| アルゴノートPIWIドメイン | |||||||||
|---|---|---|---|---|---|---|---|---|---|
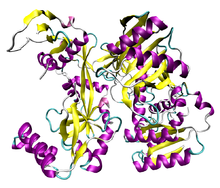 ピュロコックス・フリオスス(Pyrococcus furiosus)由来のアルゴノートタンパク質。(PDB: 1U04)。(左側) PAZドメイン (右側) PIWIドメイン | |||||||||
| 識別子 | |||||||||
| 略号 | Piwi | ||||||||
| Pfam | PF02171 | ||||||||
| InterPro | IPR003165 | ||||||||
| PROSITE | PS50822 | ||||||||
| CDD | cd02826 | ||||||||
| |||||||||
| アルゴノートPazドメイン | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| 略号 | Paz | ||||||||
| Pfam | PF12212 | ||||||||
| InterPro | IPR021103 | ||||||||
| SCOP | b.34.14.1 | ||||||||
| SUPERFAMILY | b.34.14.1 | ||||||||
| |||||||||

(右図) 二本鎖RNAと複合体を形成したアルゴノートタンパク質のPIWIドメイン(PDB: 1YTU)。ガイド鎖上の5′塩基と保存されたチロシン残基(水色)との間の塩基スタッキング相互作用が強調されている。安定化2価カチオン(マグネシウム)は灰色の球で示されている。
RNA干渉 編集
RNA干渉(RNAi)は、RNA分子が遺伝子発現を抑制する生物学的プロセスである。抑制方法は、特定のmRNA分子を破壊したり、単純にタンパク質の翻訳を抑制することである[2]。RNA干渉は寄生ヌクレオチド配列から細胞を守る上で重要な役割を果たしている。動物を含む多くの真核生物ではRNA干渉経路が見られ、それは酵素Dicerによって開始される。Dicerは、長い二本鎖RNA分子(dsRNA、ウイルスや低分子干渉RNAに多く見られる)を、約20ヌクレオチドのsiRNAの短い二本鎖断片に切断する。次に、dsRNAはパッセンジャー鎖とガイド鎖という2つの一本鎖RNA(ssRNA)に分離される。その後、パッセンジャー鎖は分解され、ガイド鎖はRNA誘導サイレンシング複合体(RISC)に組み込まれる。RNAiの最もよく研究されている成果は、転写後遺伝子サイレンシングである。これは、ガイド鎖がメッセンジャーRNA分子内の相補的配列と対になって、RNA誘導サイレンシング複合体(RISC)の中心にあるアルゴノートによる切断を誘導することで起こる。
アルゴノートタンパク質は、RNA誘導サイレンシング複合体(RISC)の活性部分であり、結合したsiRNAに相補的な標的mRNA鎖を切断する[3]。理論的には、Dicerは短い二本鎖フラグメントを生成するので、機能的な一本鎖siRNAも2つ生成されるはずである。しかし、ここでは2つの一本鎖RNAのうち1つだけが標的mRNAとの塩基対形成に利用される。これはガイド鎖と呼ばれ、アルゴノートタンパク質に組み込まれ、遺伝子サイレンシングを引き起こす。もう一方の、パッセンジャー鎖と名づけられた一本鎖は、RNA誘導サイレンシング複合体のプロセス中に分解される[4]。
アルゴノートが低分子RNAと結合すると、PIWIドメインによって与えられる酵素活性により、低分子干渉RNAのパッセンジャー鎖のみが切断される。RNA鎖の分離とアルゴノートタンパク質への組み込みは、(非対称ルールとして知られる)RNA二重鎖の5′末端における水素結合相互作用の強さによって左右される。また、中間RNA二重鎖の相補性の程度によって、miRNAがどのように異なるタイプのアルゴノートタンパク質に振り分けられるがか決まる。
動物では、miRNAに関連するアルゴノートが、mRNAの3′非翻訳領域に結合し、さまざまな方法でタンパク質の生成を妨げる。アルゴノートタンパク質が標的mRNAに動員されると、mRNAの分解を誘発する。アルゴノート-miRNA複合体は、mRNAの5′末端にある機能的リボソームの形成にも影響を与える可能性がある。ここでの複合体は翻訳開始因子と競合し、さらに/またはリボソーム組み立てを抑止する。また、アルゴノート-miRNA複合体は、ペプチドや翻訳後修飾酵素などの細胞因子を動員してポリペプチドの成長を低下させ、タンパク質の産生を調整する[5]。
植物では、標的mRNAを用いてde novo二本鎖RNA(dsRNA)が生成されると、未知のRNase-III様の酵素が新たなsiRNAを生成し、触媒アミノ酸残基を欠いたPIWIドメインを含むアルゴノートタンパク質にロードされ、別のレベルの特異的遺伝子サイレンシングが誘導される可能性がある。
機能ドメインと機構 編集
アルゴノート(AGO)遺伝子ファミリーは、N末端(N)、Linker-1(L1)、PAZ、Linker-2(L2)、Mid、およびC末端PIWIドメインの6つの特徴的なドメインをコードする[5]。
PAZドメインの名称は、このドメインが保存されていることが最初に認識されたショウジョウバエのpiwi、シロイヌナズナのargonaute-1、シロイヌナズナのzwille(poinheadとも呼ばれ、後にargonaute-10と改名)の遺伝子名から作られた頭字語である。PAZドメインは、siRNA、miRNA、piRNAの一本鎖3′末端を、配列に依存せずに認識するRNA結合モジュールである。
ショウジョウバエのPIWIタンパク質は、この特徴的なモチーフにその名前を与えた。構造的にはRNaseHに似ており、PIWIドメインは標的の切断に不可欠である。アスパラギン酸-アスパラギン酸-グルタミン酸の三徴候がある活性部位は、触媒作用に必要な2価金属イオンを内部に持つ。進化の過程でこの保存された特徴を失ったAGOファミリーメンバーは切断活性を持たない。ヒトのAGOでは、PIWIモチーフはPIWIボックスでタンパク質間相互作用も媒介し、RNase IIIドメインの1つでDicerに結合する[6]。
PIWIドメインとMidドメインの境界には、機能的に不可欠であることがわかっているsiRNA、miRNA、piRNAの5′リン酸が存在する。Midの中にはMCモチーフがあり、これはeIF4Eに見られるキャップ結合構造モチーフを模倣するために提案されたホモログ構造である。その後、MCモチーフがmRNAのキャップ結合には関与していないことが判明した[5]。
ファミリーメンバー 編集
ヒトには、8つのAGOファミリーメンバーがあり、そのうちのいくつかは集中的に研究されている。ただし、AGO1~4はmiRNAをロードできるにもかかわらず、エンドヌクレアーゼ活性、したがってRNAi依存性の遺伝子サイレンシングはAGO2にのみ属している。PAZドメインとPIWIドメインがファミリー全体で配列保存されていることを考慮すると、AGO2の独自性は、N末端またはPAZモチーフとPIWIモチーフをつなぐ間隔領域のいずれかに起因すると推測される[6]。
植物のいくつかのAGOファミリーでも多くの研究努力が集まっている。AGO1は明らかにmiRNAと関連するRNA分解に関与しており、形態形成において中心的な役割を果たしている。いくつかの生物でそれはエピジェネティック的サイレンシングで厳密に必要とされる。それはmiRNA自身によって調節されている。AGO4は、RNAiによるRNA分解には関与しないが、small RNA(smRNA)経路を介して、DNAメチル化やその他のエピジェネティック的な調節に関与する。AGO10は、植物の発生に関与している。AGO7はAGO1および10とは異なる機能を持ち、導入遺伝子によって誘発される遺伝子サイレンシングには見られない。その代わりに、植物の発育のタイミングに関係している[7]。
疾患と治療ツール 編集
膵臓がんのように特定の同定された遺伝子の選択的な発現または発現の高進に関与する疾患の場合、RNA干渉は高い配列特異性によって適切な治療法として、特に変異した内因性遺伝子配列に関連するがんと闘うのに適している可能性がある。いくつかの非コードRNA(microRNA)がヒトのがんと関連していることが報告されており、たとえばmiR-15aやmiR-16aは、患者で頻繁に欠失や下方制御を起こしている。miRNAの生物学的機能は完全には解明されていないが、発生および代謝中の細胞増殖と細胞死の調整におけるmiRNAの役割が明らかになってる。miRNAは、さまざまなレベルで負または正の調節を行うことができると期待されており、それは特定のmiRNAと標的塩基対との相互作用およびそれらを認識する補因子に依存する[8]。
多くのウイルスがDNAではなくRNAを遺伝物質とし、ライフサイクルの中で少なくとも一度は二本鎖のRNAを作ることが広く知られていることから、RNA干渉は、ウイルスから生物を守るための潜在的で進化的に古いメカニズムである可能性があると考えられてきた。Dicerが生成する低分子干渉RNAは、エンドヌクレアーゼであるRNA誘導サイレンシング複合体(RISC)をmRNAへ導くことで、配列特異的な転写後遺伝子サイレンシングを引き起こす。このプロセスは、アカパンカビ菌(これはクエリングとして知られる)、植物(転写後遺伝子サイレンシング)、哺乳類細胞(RNAi)など、幅広い生物で見られる。低分子RNAと標的との間に完全またはほぼ完全な配列相補性がある場合、RISCのアルゴノートタンパク質成分が標的転写物の切断を仲介し、この機構は主に翻訳の抑制を伴っている。
重要なのは、アルゴノート4(AGO4)欠損インフルエンザ感染マウスは、in vivo(生体内)での負荷量やウイルス力価が著しく高く[9]、これはAGO1やAGO3欠損マウスとは対照的なことである[10]。そのため、哺乳類細胞においてAGO4の機能を特異的に促進することは、効果的な抗ウイルス戦略となる可能性がある。
原核生物アルゴノートタンパク質のバイオテクノロジーへの応用 編集
2016年、河北科学技術大学のグループが、Natronobacterium gregoryi由来の原核生物アルゴノートタンパク質を使用したゲノム編集を報告した。しかし、アルゴノートタンパク質がDNA誘導性ヌクレアーゼとしてゲノム編集に適用されているという証拠は疑問視されており、主要ジャーナルからその主張は撤回されている[11]。2017年、イリノイ大学のグループは、ピュロコックス・フリオスス(Pyrococcus furiosus)から取り出した原核生物アルゴノートタンパク質(PfAgo)をガイドDNAとともに使用して、人工制限酵素としてin vivo(試験管内)でDNAを編集したと報告した[12]。PfAgoベースの人工制限酵素はまた、酵素的ニッキングを介してネイティブDNA配列上にデータを保存するためにも使用された[13]。
脚注 編集
- ^ Bohmert K, Camus I, Bellini C, Bouchez D, Caboche M, Benning C (January 1998). “AGO1 defines a novel locus of Arabidopsis controlling leaf development”. The EMBO Journal 17 (1): 170–180. doi:10.1093/emboj/17.1.170. PMC 1170368. PMID 9427751.
- ^ Guo H, Ingolia NT, Weissman JS, Bartel DP (August 2010). “Mammalian microRNAs predominantly act to decrease target mRNA levels”. Nature 466 (7308): 835–840. Bibcode: 2010Natur.466..835G. doi:10.1038/nature09267. PMC 2990499. PMID 20703300.
- ^ Kupferschmidt K (August 2013). “A lethal dose of RNA”. Science 341 (6147): 732–733. Bibcode: 2013Sci...341..732K. doi:10.1126/science.341.6147.732. PMID 23950525.
- ^ Gregory RI, Chendrimada TP, Cooch N, Shiekhattar R (November 2005). “Human RISC couples microRNA biogenesis and posttranscriptional gene silencing”. Cell 123 (4): 631–640. doi:10.1016/j.cell.2005.10.022. PMID 16271387.
- ^ a b c Hutvagner G, Simard MJ (January 2008). “Argonaute proteins: key players in RNA silencing”. Nature Reviews. Molecular Cell Biology 9 (1): 22–32. doi:10.1038/nrm2321. hdl:10453/15429. PMID 18073770.
- ^ a b Meister G, Landthaler M, Patkaniowska A, Dorsett Y, Teng G, Tuschl T (July 2004). “Human Argonaute2 mediates RNA cleavage targeted by miRNAs and siRNAs”. Molecular Cell 15 (2): 185–197. doi:10.1016/j.molcel.2004.07.007. PMID 15260970.
- ^ Meins F, Si-Ammour A, Blevins T (2005). “RNA silencing systems and their relevance to plant development”. Annual Review of Cell and Developmental Biology 21 (1): 297–318. doi:10.1146/annurev.cellbio.21.122303.114706. PMID 16212497.
- ^ Hannon GJ (July 2002). “RNA interference”. Nature 418 (6894): 244–251. Bibcode: 2002Natur.418..244H. doi:10.1038/418244a. PMID 12110901.
- ^ Adiliaghdam, F., Basavappa, M., Saunders, T. L., Harjanto, D., Prior, J. T., Cronkite, D. A., ... & Jeffrey, K. L. (2020). A Requirement for Argonaute 4 in Mammalian Antiviral Defense. Cell reports, 30(6), 1690-1701. doi:10.1016/j.celrep.2020.01.021 PMC 7039342 PMID 32049003
- ^ Van Stry, M., Oguin, T. H., Cheloufi, S., Vogel, P., Watanabe, M., Pillai, M. R., ... & Bix, M. (2012). Enhanced susceptibility of Ago1/3 double-null mice to influenza A virus infection. Journal of virology, 86(8), 4151-4157. doi:10.1128/JVI.05303-11 PMC 3318639 PMID 22318144
- ^ Cyranoski, David (2017). “Authors retract controversial NgAgo gene-editing study”. Nature. doi:10.1038/nature.2017.22412.
- ^ Enghiad B, Zhao H (May 2017). “Programmable DNA-Guided Artificial Restriction Enzymes”. ACS Synthetic Biology 6 (5): 752–757. doi:10.1021/acssynbio.6b00324. PMID 28165224.
- ^ Tabatabaei, S. Kasra; Wang, Boya; Athreya, Nagendra Bala Murali; Enghiad, Behnam; Hernandez, Alvaro Gonzalo; Fields, Christopher J.; Leburton, Jean-Pierre; Soloveichik, David et al. (8 April 2020). “DNA punch cards for storing data on native DNA sequences via enzymatic nicking”. Nature Communications 11 (1): 1–10. doi:10.1038/s41467-020-15588-z. PMC 7142088. PMID 32269230.