ヌクレアーゼ
ヌクレアーゼ(英: nuclease)は、核酸のヌクレオチド間のホスホジエステル結合を切断することができる酵素である。古くはヌクレオデポリメラーゼ(英: nucleodepolymerase)またはポリヌクレオチダーゼ(英: polynucleotidase)とも呼ばれた。ヌクレアーゼは、標的分子の一本鎖および二本鎖をさまざまな様式で切断する。ヌクレアーゼは、生体内におけるDNA修復に関わる多くの側面で不可欠な機構である。ある種のヌクレアーゼの欠陥は、遺伝的不安定性や免疫不全を引き起こす可能性がある[1]。また、ヌクレアーゼは、分子クローニングにも広く用いられている[2]。
ヌクレアーゼは、その活性座位によって2種類に大別される。エキソヌクレアーゼ (en:英語版) は、核酸を末端から消化する。エンドヌクレアーゼ (en:英語版) は、標的分子の途中にある領域に作用する。これらはさらに、デオキシリボヌクレアーゼとリボヌクレアーゼに分類できる。前者はDNAに作用し、後者はRNAに作用する[2]。
歴史 編集
1960年代後半、科学者のスチュアート・リンとヴェルナー・アーバーは、大腸菌(E. coli)でのファージ増殖制限に関与する2種類の酵素の標本を単離した[3][4]。これらの酵素の一方はDNAにメチル基を付加してメチル化DNAを生成し、他方は非メチル化DNAを分子の長さ方向に沿ったさまざまな場所で切断した。最初の種類の酵素は「メチラーゼ」、もう一方は「制限ヌクレアーゼ」と呼ばれた。DNA分子を「切り貼り」するための必要な道具を集めていた科学者にとって、これらの酵素学的な道具は重要であった。そこで必要とされたのが、分子の長さに沿ったランダムな位置ではなく、特定の部位でDNAを切断し、科学者が予測可能かつ再現性のある方法でDNA分子を切断できる道具であった。
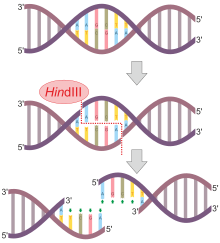
1968年、ジョンズ・ホプキンス大学で研究していたハミルトン・スミス、K・W・ウィルコックス、トーマス・J・ケリーが、特定のDNAヌクレオチド配列に依存して機能する最初の制限酵素を単離し、その特徴を明らかにしたとき、重要な進展がもたらされた。このグループは、インフルエンザ菌を使った研究で、6つの塩基対からなる特定の配列内の特定の位置で常にDNA分子を切断するHindIIと呼ばれる酵素を単離した。彼らは、HindII酵素が常にその配列の中心(3番目と4番目の塩基対の間)を直接切断することを発見した。
数値分類体系 編集
ほとんどのヌクレアーゼは、「国際生化学・分子生物学連合命名法委員会」のEC番号によって、加水分解酵素(ヒドロラーゼ)(EC番号3)として分類されている。ヌクレアーゼは、ホスホジエステラーゼ、リパーゼ、およびホスファターゼと同様に、加水分解酵素のサブグループであるエステラーゼ(EC番号3.1)に属する。ヌクレアーゼが属するエステラーゼは、EC番号3.1.11 - EC番号3.1.31に分類される。
構造 編集
ヌクレアーゼの一次構造は、最小限の活性部位が保存される他は大部分が保存されておらず、その表面は主に、酸性および塩基性のアミノ酸残基から構成されている。ヌクレアーゼはフォールディングファミリーに分類される[5]。
部位認識 編集
ヌクレアーゼは、核酸分子を切断する前に、核酸と結合しなければならない。それには、ある程度の認識が必要となる。ヌクレアーゼは、その認識と結合の様式において、特異的および非特異的な結合の両方をさまざまに用いる。どちらの様式も、生存生物内、特にDNA修復において重要な役割を果たしている[7]。
DNA修復に関与する非特異的エンドヌクレアーゼは、DNAを走査して標的配列や損傷を探し出すことができる。このようなヌクレアーゼは、DNAに沿って拡散し、標的に遭遇すると、その活性部位の残基はDNAの化学基と相互作用する。EcoRV、BamHI、PvuIIなどのエンドヌクレアーゼの場合、この非特異的結合には、タンパク質の最小表面積とDNAとの間の静電的相互作用が含まれる。この弱い結合により、DNAの全体的な形状は変形せず、B型構造のままとなる[7]。
これに対し、部位特異的ヌクレアーゼは、はるかに強い結合を形成し、DNAをそのDNA結合ドメインの深い溝に引き込む。これによりDNAの三次構造が大きく変形し、塩基性(正電荷を持つ)の残基を多く含む表面が実現される。それはDNAとの広範な静電的相互作用に関与する[7]。
DNA修復に関与するヌクレアーゼの中には、部分的な配列特異性を示すものがある。ただし、ほとんどは非特異的で、塩基対の不一致によってDNA骨格に生じた構造異常を認識する[7]。
構造特異的ヌクレアーゼ 編集
詳細については、フラップエンドヌクレアーゼを参照。
配列特異的ヌクレアーゼ 編集
| 酵素 | 起源 | 認識する配列 | 切断位置 |
|---|---|---|---|
| HindII | インフルエンザ菌 (Haemophilus influenzae) |
5'–GTYRAC–3' |
5'–GTY RAC–3' |
| R = A or G; Y = C or T | |||
HindIIの発見以来、230以上の菌株から900種類以上の制限酵素が単離され、中には配列特異的なものも、そうではないものもある。これらの制限酵素には、一般にその起源を反映した名前がつけられる。名前の最初の文字は属名に由来し、次の2文字は、それらが単離された原核細胞の種名に由来する。たとえば、EcoRIはEscherichia coli RY13(大腸菌 RY13株)に由来し、HindIIはHaemophilus influenzae strain Rd(インフルエンザ菌 Rd株)に由来する。ヌクレアーゼ名に続く数字は、その酵素が単一菌株から単離された順序を示す(例:EcoRI、EcoRII)。
エンドヌクレアーゼ 編集
制限エンドヌクレアーゼは、DNA分子を長さ方向に沿って「走査」することで機能を果たす。それは、特定の認識配列に遭遇するとDNA分子に結合し、2つの糖-リン酸主鎖をそれぞれ1つずつ切断する。この2つの切断の位置は互いに関連し、また認識配列自体にも関連しており、制限エンドヌクレアーゼの独自性によって決定される。エンドヌクレアーゼが異なれば切断の仕方も異なるが、特定のエンドヌクレアーゼは、作用するDNA分子に関わらず、特定の塩基配列を常に同じように切断する。切断が行われると、DNA分子は断片に分割される。
スタガード・カット 編集
すべての制限エンドヌクレアーゼが、上記のHindIIのように対称的に切断し、平滑末端を残すわけではない。多くのエンドヌクレアーゼは、互いに正対しない位置でDNA主鎖を切断し、オーバーハングを形成する。たとえば、ヌクレアーゼEcoRIの認識配列は5'—GAATTC—3'である。
| 酵素 | 起源 | 認識する配列 | 切断位置 |
|---|---|---|---|
| HindIII | インフルエンザ菌 (Haemophilus influenzae) |
3'–TTCGAA–5' |
3'–TTCGA A–5' |
| EcoRI | 大腸菌 (Escherichia coli) |
3'–CTTAAG–5' |
3'–CTTAA G–5' |
| BamHI | バチルス・アミロリケファシエンス (Bacillus amyloliquefaciens) |
3'–CCTAGG–5' |
3'–CCTAG G–5' |
酵素はこの配列に遭遇すると、Gと最も近いA塩基残基の間の各骨格を切断する。切断されて生成した断片は、互いに相補的な塩基を接続する比較的弱い水素結合によってのみ保持される。この結合の弱さは、DNA断片が互いに分離することを可能にする。得られた各断片には、不対塩基からなる突出した5'末端がある。別の酵素は、DNA骨格に切れ目を作り、3' 末端が突出する。突出した末端(3' と 5' ともに)は、塩基の相補的な配列と結合する傾向があり、そのために「粘着末端」と呼ばれることもある。言い換えると、不対配列5'—AATT—3'が別の不対配列3'—TTAA—5'に遭遇すると、互いに結合する。これらは互いに粘着性を持つ。次にリガーゼと呼ばれる酵素が用いられて、2つの分子のリン酸骨格を結合する。粘着末端の細胞由来、あるいは種由来でさえ、その粘着性に影響を与えない。たとえ配列の一方がヒトDNAに由来し、もう一方が細菌DNAに由来する場合でも、相補的配列の任意の組は結合する傾向がある。実際、この粘着性の性質によって、異なる起源のDNAから構成される組換えDNA分子の作成が可能となり、遺伝子工学技術が誕生した。
自然界での役割 編集
DNA修復 編集
すべての細胞は遺伝情報の媒体としてDNAに依存しているため、遺伝子の品質管理はすべての生物にとって不可欠な機能である。DNAの複製は誤りが起こりやすい過程であり、DNA分子自体も、多くの代謝的あるいは環境的要因のストレスを受けやすい。身近な例として、活性酸素種、近紫外線、あるいは電離放射線があげられる。多くのヌクレアーゼは、損傷部位を認識し、それを周囲のDNAから切断することでDNA修復に関与する。これらの酵素は単独で、あるいは複合体として機能する。DNA修復に関与するほとんどのヌクレアーゼは配列特異的ではない。これらは二本鎖DNA(dsDNA)の二次構造の変形を通じて損傷部位を認識する[5]。
複製校正 編集
DNAの複製中に、DNAポリメラーゼは、相補的な鋳型鎖に対して新しいDNA鎖を伸長させる。ほとんどのDNAポリメラーゼは、ポリメラーゼと校正エキソヌクレアーゼという2つの異なる酵素的なドメインから構成されている。ポリメラーゼは、新しい鎖を 5'→3' 方向に伸長させる。エキソヌクレアーゼは、同じ鎖から誤ったヌクレオチドを 3'→5' 方向に除去する。このエキソヌクレアーゼ活性は、DNAポリメラーゼが校正を行うために不可欠である。これらのヌクレアーゼを不活性化したり失ったりする欠失は、影響を受けた微生物の突然変異率や死亡率を増加させ、マウスでは発癌率を増加させる[8]。
複製フォークの停止 編集
DNA損傷の多くの形態は複製フォークの進行を停止させ、DNAポリメラーゼや関連する仕組みがそのフォークを放棄する原因となる。その時、これはフォーク固有のタンパク質によって処理する必要がある。最も有名な酵素はMUS81である。酵母において、その欠失は減数分裂の欠陥に加え、紫外線またはメチル化損傷感受性を引き起こす[5]。
岡崎フラグメント処理 編集
細胞内では、岡崎フラグメントRNAプライマーを複製から除去する仕事が広範に行われている。このようなプライマーの多くは、RNase Hファミリーのエンドヌクレアーゼによって、新たに合成されたラギング鎖DNAから切り出される。また、真核生物や古細菌では、フラップエンドヌクレアーゼFEN1も岡崎フラグメントの処理に関与している[5]。
ミスマッチ修復 編集
既知の生物におけるDNAミスマッチ修復は、一連のミスマッチ特異的エンドヌクレアーゼによって行われる。原核生物では、この役割は主にMutSLHと超短鎖パッチ修復関連タンパク質によって担われている。
MutSLHシステム(MutS、MutL、MutHから構成される)は、点変異と小ターン変異を修正する。MutSはミスマッチを認識して結合し、ここでMutLとMutHを動員する。MutLは、MutSとMutHの相互作用を仲介し、MutHのエンドヌクレアーゼ活性を高める。MutHは、ヘミメチル化された5'—GATC—3'部位を認識し、非メチル化鎖(最近合成された鎖)のGの隣で切断する。
VSP修復はエンドヌクレアーゼVsrによって開始される。Vsrは、メチル化シトシンのチミンへの自発的な脱アミノ化によって起こる特定のT/Gミスマッチを修正する。Vsrは配列5'—CTWGG—3'を認識し、ミスマッチしたチミンの 5' 側のDNA鎖にニック(切れ目)を入れる(前出の配列で下線が引かれている)。次に、エキソヌクレアーゼRecJ、ExoVII、またはExoIのいずれかが、DNAポリメラーゼが鎖のギャップを再合成する前に、この部位を分解する[5]。
塩基除去修復 編集
AP部位の形成は、dsDNAでよく見られる。これは、塩基除去修復の中間段階として、自然加水分解とDNAグリコシラーゼの活性によって生じる。これらのAP部位は、APエンドヌクレアーゼによって除去され、その部位周辺の一本鎖切断を引き起こす[5]。
ヌクレオチド除去修復 編集
ヌクレオチド除去修復は、損傷したヌクレオチドを除去し置き換えることである。これを前述の塩基除去修復と混同してはならない。架橋、付加体、および損傷(紫外線や活性酸素によって生じる)の例は、その修復経路の引き金となりうる。そのような損傷を受けたヌクレオチドを含む一本鎖DNAの短い区間は、損傷個所の上流と下流にニックを入れる別々のエンドヌクレアーゼによって二重鎖DNAから除去される。これらのヌクレアーゼに影響を与える欠失や突然変異は、紫外線による損傷あるいは発癌に対する感受性を高める。このような異常は、神経の発達にさえ影響を与えることもある。
細菌では、UvrB-UvrC複合体によって両方の切断が行なわる。出芽酵母では、Rad2とRad1-Rad10複合体がそれぞれ 5' 切断と 3' 切断が行われる。哺乳類では、同族体のXPGとXPF-ERCC1が同じようにそれぞれのニックに作用する[9]。
二本鎖切断修復 編集
細胞内では、二重鎖切断が、意図的または非意図的にかかわらず定期的に行われる。意図的でない切断は、通常、電離放射線、さまざまな外因性や内因性の化学物質、および複製フォークの停止によって発生する。意図的な切断は、減数分裂やV(D)J組換えの中間体として生成され、主に相同組換えや非相同末端結合によって修復される。どちらの場合も、修復が行われる前に、二本鎖切断の末端がヌクレアーゼによって処理される必要がある。そのようなヌクレアーゼの一つに、Rad50と複合体化したMre11がある。Mre11の変異は、毛細血管拡張性運動失調症様疾患を引き起こすことがある[9]。
V(D)J組換えでは、二本鎖切断に関連するステムループ構造を開き、その後に両端を結合させることが含まれる。アルテミス-DNAPKcs複合体がこの反応に関与している。アルテミスは単独でも 5' → 3' ssDNAエキソヌクレアーゼ活性を示すが、DNA-PKcsとの複合体形成により、ステムループをエンドヌクレアーゼ的に処理することが可能となる。いずれかのタンパク質の欠陥は、重度の免疫不全を引き起こす[9]。
一方、相同組換えには、Dループまたはホリデイジャンクションで接続された2つの相同DNA二重鎖 (en:英語版) が含まれる。細菌では、RuvCのようなエンドヌクレアーゼが、ジャンクション中心近くの2つの対称的な部位でジャンクションを切断することにより、ホリデイジャンクションを2つの別々のdsDNAに分解する。真核生物では、FEN1、XPF-ERCC1、およびMUS81がDループを切断し、Cce1/Ydc2がミトコンドリア内でホリデイジャンクションを処理する[9]。
メガヌクレアーゼ 編集
特定のヌクレアーゼが特定のDNA分子を切断する頻度は、DNAの複雑さとヌクレアーゼの認識配列の長さに依存する。偶然に特定の順序で塩基が見つかる統計的可能性のため、認識配列が長くなると切断の頻度は低くなる。たとえば、特定の4塩基配列(仮想的なヌクレアーゼの認識部位に相当)は、平均して256塩基対ごとに出現すると予測されるが(4^4=256)、任意の6塩基配列は平均して4096塩基対ごとに1回出現すると予測される(4^6=4096)。
ヌクレアーゼの独特なファミリーの1つにメガヌクレアーゼがある。メガヌクレアーゼは、12-40塩基対からなる、より大きな認識配列(したがってあまり一般的ではない)を持つことが特徴である。これらのヌクレアーゼは、植物や哺乳類などの複雑な生物における遺伝子工学やゲノム工学への応用に特に有用である。一般に、数十億塩基対に及ぶ大きなゲノムは、従来のヌクレアーゼでは頻繁で有害な部位特異的切断を引き起こす。
参照項目 編集
- 制限酵素 - DNA分子内の特定の認識部位またはその近くでDNAを特異的に切断する酵素(制限エンドヌクレアーゼの一種)
- HindIII - ヘモフィルス-インフルエンザ菌から単離されたデオキシヌクレアーゼ制限酵素
- リガーゼ - 新しい化学結合を形成して2つの大きな分子を結合できる酵素
- 小球菌ヌクレアーゼ - 一本鎖の核酸を優先的に消化するエンド-エキソヌクレアーゼ
- ヌクレアーゼプロテクションアッセイ - 細胞から抽出した不均一なRNA試料中の個々のRNA分子を同定するための実験方法
- ヌクレアーゼS1 - 一本鎖 DNA (ssDNA) および RNA をオリゴモノヌクレオチドまたはモノヌクレオチドに分割するエンドヌクレアーゼ酵素
- PINドメイン - 一本鎖RNAを切断するヌクレアーゼ酵素として機能するタンパク質ドメイン
- ポリメラーゼ - 高分子や核酸の長鎖を合成する酵素
脚注 編集
- ^ Nishino T, Morikawa K (December 2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. Oncogene 21 (58): 9022–32. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ^ a b Rittié L, Perbal B (June 2008). “Enzymes used in molecular biology: a useful guide”. Journal of Cell Communication and Signaling 2 (1–2): 25–45. doi:10.1007/s12079-008-0026-2. PMC 2570007. PMID 18766469.
- ^ Linn S, Arber W (April 1968). “Host specificity of DNA produced by Escherichia coli, X. In vitro restriction of phage fd replicative form”. Proceedings of the National Academy of Sciences of the United States of America 59 (4): 1300–6. Bibcode: 1968PNAS...59.1300L. doi:10.1073/pnas.59.4.1300. PMC 224867. PMID 4870862.
- ^ Arber W, Linn S (1969). “DNA modification and restriction”. Annual Review of Biochemistry 38: 467–500. doi:10.1146/annurev.bi.38.070169.002343. PMID 4897066.
- ^ a b c d e f Nishino T, Morikawa K (December 2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. Oncogene 21 (58): 9022–32. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ^ Winkler FK, Banner DW, Oefner C, Tsernoglou D, Brown RS, Heathman SP, Bryan RK, Martin PD, Petratos K, Wilson KS (May 1993). “The crystal structure of EcoRV endonuclease and of its complexes with cognate and non-cognate DNA fragments”. The EMBO Journal 12 (5): 1781–95. doi:10.2210/pdb4rve/pdb. PMC 413397. PMID 8491171.
- ^ a b c d Nishino T, Morikawa K (December 2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. Oncogene 21 (58): 9022–32. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ^ Nishino T, Morikawa K (December 2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. Oncogene 21 (58): 9022–32. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ^ a b c d Nishino T, Morikawa K (December 2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. Oncogene 21 (58): 9022–32. doi:10.1038/sj.onc.1206135. PMID 12483517.