核膜孔
核膜孔(かくまくこう、英:Nuclear pore)とは、核の内外を連絡する穴である。真核生物の核膜の内膜と外膜が融合する場にあり、核と細胞質間の物質の移動はこの核膜孔を介して行われる。核膜孔には核膜孔複合体が位置し、これは8個のサブユニットが回転対称に配置された巨大なタンパク質複合体である。脊椎動物の細胞の核膜には約1000個の核膜孔複合体が存在するが、その数は細胞種や生活環の段階に依存して変動する[1]。

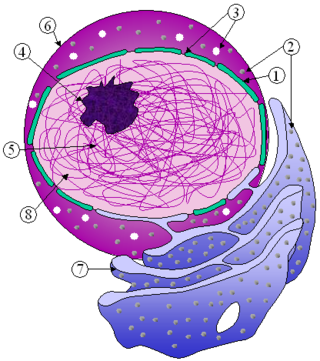
核から細胞質への移動にはRNAやリボソームタンパク質が、細胞質から核内への移動には核タンパク質 (DNAポリメラーゼやラミンなど)、炭水化物、シグナル伝達物質と脂質が含まれる。より小さな分子は孔を通って単純拡散するが、より大きな分子は特定のシグナル配列によって認識され、ヌクレオポリンと呼ばれる核膜孔複合体構成タンパク質の助けによって、核内または核外へ拡散する。各核膜孔複合体が毎秒1000個の輸送を行うことができることは特筆に値する[2]。ヌクレオポリンを介した輸送は直接的にはエネルギーを必要としないが、RANサイクルに関連した濃度勾配に依存する。
構造 編集
核膜孔の数は細胞の状態によって様々だが、1核あたり出芽酵母では平均100個強 (約10個/μm2)[3] 、ラットNRK細胞では1700個前後 (約3個/μm2)[4] 、という報告がある。
ヒトの核膜孔複合体は約 110 MDaの構造である。核膜孔複合体を作り上げるタンパク質はヌクレオポリン(nucleoporin, Nup) として知られる。核膜孔複合体は少なくとも456個のタンパク質分子からなり、34種類の異なるタンパク質(ヌクレオポリン)を含む[5]。典型的にはヌクレオポリンの約半数はソレノイド状のタンパク質ドメイン――α-ソレノイドまたはβ-プロペラを持っており、その両方を別々の構造ドメインとして含んでいることもある。残りの半数は、典型的な天然変性タンパク質の構造的特徴を示す非常に柔軟なタンパク質で、安定な立体構造を持たない[6]。これらのタンパク質はFGヌクレオポリンと呼ばれ、その名はそのアミノ酸配列にはフェニルアラニンーグリシンの反復配列が多数存在することに由来する[7]。
核膜孔複合体全体の直径は、脊椎動物では約 120 nmである[8]。チャネルの直径はヒトでは 5.2 nm[9]、アフリカツメガエルでは 10.7 nmと差があり、深さは約 45 nmである[10]。一本鎖mRNAの厚さは 0.5 – 1 nmである。哺乳類の核膜孔複合体は約 124 MDaで[11]約30種類のタンパク質から構成され、それぞれが複数コピー存在する[12]。対照的に、酵母 Saccharomyces cerevisiae のものは小さく、わずか 66 MDaである[13]。
核膜孔複合体は8個のサブユニットが回転対称に配置された巨大なタンパク質複合体である。細胞質側には細胞質フィラメント、核質側には核バスケットと呼ばれる構造が突き出ていて、輸送過程ではこれらの構造と輸送される物質の相互作用が重要な役割を果たしている。実際の孔 (アウターリング) を囲む8つのタンパク質サブユニットのそれぞれから、スポーク型のタンパク質が孔へ向かって飛び出している[14]。中心部にはplugと呼ばれる構造が認められるが[14]、多様な外見を成し、カルシウム濃度と構造変化の関連が指摘されているものの詳細な役割は不明となっている。
輸送 編集
小さな粒子 (< ~30–60 kDa) は受動拡散によって核膜孔複合体を通過することができる[15][16]。より大きな粒子であっても受動拡散によって通過することはできるが、その速度は分子量に応じて徐々に低下していく。効率的な複合体の通過にはいくつかのタンパク質因子が必要であり[17]、特に核輸送受容体は、積み荷分子に結合して核膜孔複合体を経由した核内への通過(インポーチン)、核外への通過(エクスポーチン)を媒介する。核輸送受容体の最大のファミリーはカリオフェリンであり、数十種類のインポーチンとエクスポーチンが含まれる。このファミリーはさらにカリオフェリン-αとカリオフェリン-βサブファミリーに分類される。他の核輸送受容体にはNTF2やNTF2様タンパク質などがある。
輸送のメカニズムを説明するため、3つのモデルが提案されている。
タンパク質の核内輸送 編集
核局在化シグナル/配列 (nuclear localization signal/seuence, NLS) が露出している積み荷タンパク質は、迅速かつ効率的に孔を通って目的地へ送られる。NLSの配列はいくつか知られており、一般的には PKKKRKV のような塩基性残基からなる保存された配列を含んでいる[19]。NLSを有する物質はインポーチンによって核へ取り込まれる。
NLSを持つタンパク質の輸送の典型的なスキームは、まずインポーチンαがNLS配列に結合することから始まる。インポーチンαはインポーチンβが結合するためのブリッジとして機能する。インポーチンβ-インポーチンα-積み荷タンパク質複合体は核膜孔を通過して拡散する。いったん複合体が核内へ入ると、GTP結合型Ran (Ran-GTP) がインポーチンβに結合し、インポーチンβを複合体から解離させる。その後、cellular apoptosis susceptibility protein (CAS) という核内でRan-GTPと結合するエクスポーチンがインポーチンαを積み荷タンパク質から解離させる。インポーチンβ-RanGTP複合体とインポーチンα-CAS-RanGTP複合体は拡散によって細胞質へ送り返される。細胞質ではRanに結合したGTPがGDPへ加水分解され、それに伴ってインポーチンβとインポーチンαが解離し、新たなNLSタンパク質の輸送のラウンドが可能になる[20]。
積み荷タンパク質はシャペロンタンパク質の助けを借りて孔を通過するが、孔の通過自体はエネルギー依存的ではない。しかし、核内輸送サイクル全体としては、2分子のGTPの加水分解が必要であり、エネルギー依存的な能動輸送であると見なされる。核内輸送のサイクルは、RanGTPの核-細胞質間濃度勾配によって駆動される。この勾配は、Ran分子のGDPをGTPに交換するタンパク質であるRanGEFが核のみに局在していることで形成される。RanGEFの局在のために、核内のRan-GTPの濃度は細胞質と比較して高くなっている。
核外輸送 編集
リボソームのサブユニットやmRNAなどの一部の分子や高分子複合体は、核から細胞質へ輸送される必要がある。核外輸送は核内輸送と似たメカニズムで行われる。
典型的な核外輸送スキームでは、核外搬出シグナル (nuclear export signal/sequence, NES) を持つタンパク質は、核内でエクスポーチン (CRM1など)、Ran-GTPとヘテロ三量体複合体を形成する。その後複合体は細胞質へ拡散し、GTPが加水分解されてNESタンパク質が遊離する。CRM1-RanGDPは拡散によって核内へ戻り、そこでRanGEFによってGDPがGTPへ交換される。この過程も1分子のGTPを消費するためエネルギー依存的である。エクスポーチンCRM1による核外輸送は、レプトマイシンBによって阻害される[21]。
RNAの核外輸送 編集
RNAの核外輸送には、RNAの種類ごとにさまざまな経路が存在する。RNAの核外輸送は、輸送に関与するRNA結合タンパク質に存在するNESのシグナルによって媒介される (ただしアダプタータンパク質を持たないtRNAは除く)。mRNA以外の細胞のRNA (tRNA、rRNA、U snRNA、miRNA) やウイルスRNAの輸送はRanGTPに依存する[22]。mRNAの核外輸送には、保存されたmRNA核外輸送因子が必要である。核外輸送因子はMex67/Tap (大サブユニット) とMtr2/p15 (小サブユニット) である。高等生物では、mRNAの核外輸送はスプライシング依存的であると考えられており、TREXと呼ばれるタンパク質複合体がスプライシングを受けたmRNAへリクルートされる[23]。RNA結合能が非常に弱いTAPに対するアダプターとしてTREXは機能する。ヒストンのような特別なmRNAには、スプライシングに依存しない代替的なmRNA核外輸送経路が存在する。近年の研究結果から、分泌タンパク質やミトコンドリアのタンパク質をコードする遺伝子の転写産物に関しては、スプライシング依存的な核外輸送経路と代替的な経路との連携が行われていることが示唆されている[24]。
構築 編集
核膜孔複合体はゲノムへのアクセスを制御しているため、大量の転写が必要な細胞周期のステージでは、大量の核膜孔複合体が必要である。例えば、哺乳類や酵母の細胞では、核膜孔複合体の数は細胞周期のG1期からG2期の間に倍増し、卵母細胞では、発達の初期段階の迅速な有糸分裂に備えて多数の核膜孔複合体が蓄積している。間期の細胞でも、核膜孔複合体の一部は損傷を受けるため、レベルを一定に保つためには核膜孔複合体を作り続ける必要がある。一部の細胞では、転写需要の増加によって核膜孔複合体の数が増加することもある[25]。
組み立ての理論 編集
核膜孔複合体がどのように組み立てられるかについては、いくつかの仮説が存在する[26]。Nup107-160複合体のような特定のタンパク質複合体の免疫除去を行うと孔のない核が形成されるため[27]、Nup複合体は核膜の外膜と内膜の融合に関与しており、膜の融合が孔の形成の開始段階ではないと考えられる。主要なモデルは次のようなものである。
- 1つの可能性は、1つのタンパク質複合体がクロマチンに結合するものである。その後、複合体はクロマチンに近接した二重膜に挿入され、膜の融合が引き起こされる。このタンパク質複合体の周辺に他の因子が次第に結合し、核膜孔複合体が形成される。有糸分裂後の細胞では膜が最初に形成され、その後に孔が挿入される。
- 別のモデルでは、単一のタンパク質複合体ではなくpreporeが最初に形成されるとされる。このpreporeはいくつかのNup複合体が集まってクロマチンに結合することで形成される。有糸分裂後の膜の再形成の際、このpreporeの周囲に二重膜が形成される。電子顕微鏡によって、preporeと思われる構造体が核膜が形成される前のクロマチン上に観察されている[28]。細胞周期の間期には、preporeの形成は核の内部で起こる。各構成要素は既に存在する核膜孔複合体を通って輸送される。これらのNupは細胞質で合成されるとインポーチンに結合し、細胞質でのpreporeの形成が防がれでいる。核内へ輸送されると、Ran-GTPがインポーチンに結合してNupが放出され、preporeを形成できるようになる。少なくともNup107とNup153はインポーチンに結合して核内へ移行することが示されている[25]。核膜孔複合体の組み立ては未解明の中間状態がある非常に速い過程で、段階的に進行することが示唆されている[29]。
解体 編集
有糸分裂の期間中に、核膜孔複合体は段階的に解体されるように見える。核膜孔複合体のの解体は、まず周縁部に位置するNup98の解離を契機として開始され、核膜孔複合体の核を形成する足場タンパク質に先駆けて、周縁部のサブユニットの解離が起こるようである[30]。この周縁部の解体は大部分がリン酸化によって駆動されるものであると考えられている[31]。この部分的な解体に伴う構造の変化によって核膜孔複合体の透過性が増加し、細胞質のチューブリンなどの核膜の解体に関与するタンパク質や、有糸分裂の調節因子が核内に進入することが可能になる。糸状菌 Aspergillus nidulans のような semi-open型有糸分裂 (核膜の完全な解体は起こらないが、核膜孔の変化は起こる) を行う生物では、NIMAやCdk1キナーゼの活性化によって30種類のヌクレオポリンのうち14種類が核となる足場構造から解離し、核膜孔が広く開き、有糸分裂調節因子が核内に進入する[32][33][34]。完全なclosed型の有糸分裂を行う生物でも、有糸分裂期に核膜孔の透過性の変化が起こるが、その機構は不明である[35]。
出典 編集
- ^ Maul, Gerd G; Deaven, Larry (1977). “Quantitative Determination of Nuclear Pore Complexes in Cycling Cells with Differing DNA Content”. Journal of Cell Biology 73 (3): 748–760. doi:10.1083/jcb.73.3.748. PMC 2111421. PMID 406262 2014年12月12日閲覧。.
- ^ The nucleolus. Olson, Mark O. J.. Georgetown, Tex.: Landes Bioscience / Eurekah.Com. (2004). ISBN 0306478730. OCLC 54279897
- ^ Winey, M.; Yarar, D.; Giddings, T. H.; Mastronarde, D. N. (1997-11). “Nuclear pore complex number and distribution throughout the Saccharomyces cerevisiae cell cycle by three-dimensional reconstruction from electron micrographs of nuclear envelopes”. Molecular Biology of the Cell 8 (11): 2119–2132. ISSN 1059-1524. PMC 25696. PMID 9362057.
- ^ Daigle, N.; Beaudouin, J.; Hartnell, L.; Imreh, G.; Hallberg, E.; Lippincott-Schwartz, J.; Ellenberg, J. (2001-07-09). “Nuclear pore complexes form immobile networks and have a very low turnover in live mammalian cells”. The Journal of Cell Biology 154 (1): 71–84. doi:10.1083/jcb.200101089. ISSN 0021-9525. PMC 2196857. PMID 11448991.
- ^ Lin, D. H., Stuwe, T., Schilbach, S., Rundlet, E. J., Perriches, T., Mobbs, G., … Hoelz, A. (2016). Architecture of the nuclear pore complex symmetric core. Science, 352(6283), aaf1015. https://doi.org/10.1126/science.aaf1015
- ^ “Disorder in the nuclear pore complex: The FG repeat regions of nucleoporins are natively unfolded”. Proc Natl Acad Sci USA 100 (5): 2450–5. (2003). doi:10.1073/pnas.0437902100. PMC 151361. PMID 12604785.
- ^ Peters R (2006). Introduction to nucleocytoplasmic transport: molecules and mechanisms. Methods in Molecular Biology™. 322. 235–58. doi:10.1007/978-1-59745-000-3_17. ISBN 978-1-58829-362-6. PMID 16739728
- ^ Winey, Mark; Yarar, Defne; Giddings Jr., Thomas H; Mastronarde, David N (1 November 1997). “Nuclear Pore Complex Number and Distribution throughout the Saccharomyces cerevisiae Cell Cycle by Three-Dimensional Reconstruction from Electron Micrographs of Nuclear Envelopes”. Molecular Biology of the Cell 8 (11): 2119–2132. doi:10.1091/mbc.8.11.2119. PMC 25696. PMID 9362057 2014年12月12日閲覧。.
- ^ Mohr, Dagmar; Frey, Steffen; Fischer, Torsten; Güttler, Thomas; Görlich, Dirk (13 August 2009). “Characterisation of the passive permeability barrier of nuclear pore complexes”. The EMBO Journal 28 (17): 2541–2553. doi:10.1038/emboj.2009.200. PMC 2728435. PMID 19680228 2014年12月12日閲覧。.
- ^ Keminer, Oliver; Peters, Reiner (July 1999). “Permeability of Single Nuclear Pores”. Biophysical Journal 77 (1): 217–228. doi:10.1016/S0006-3495(99)76883-9. PMC 1300323. PMID 10388751.
- ^ Reichelt, R; Holzenburg, A; Buhle Jr., E L; Jarnik, M; Engel, A; Aebi, U (1 April 1990). “Correlation between Structure and Mass Distribution of the Nuclear Pore Complex and of Distinct Pore Complex Components”. Journal of Cell Biology 110 (4): 883–894. doi:10.1083/jcb.110.4.883 2014年12月12日閲覧。.
- ^ Alber, Frank; Dokudovskaya, Svetlana; Veenhoff, Liesbeth M.; Zhang, Wenzhu; Kipper, Julia; Devos, Damien; Suprapto, Adisetyantari; Karni-Schmidt, Orit et al. (29 November 2007). “Determining the architectures of macromolecular assemblies”. Nature 450 (7170): 683–694. doi:10.1038/nature06404. PMID 18046405 2014年12月12日閲覧。.
- ^ “Isolation of the yeast nuclear pore complex”. J. Cell Biol. 123 (4): 771–83. (November 1993). doi:10.1083/jcb.123.4.771. PMC 2200146. PMID 8227139.
- ^ a b Unwin, P. N.; Milligan, R. A. (1982-4). “A large particle associated with the perimeter of the nuclear pore complex”. The Journal of Cell Biology 93 (1): 63–75. ISSN 0021-9525. PMC 2112107. PMID 7068761.
- ^ “Nuclear export of RNA”. Biology of the Cell 96 (8): 639–55. (3 August 2004). doi:10.1016/j.biolcel.2004.04.014. PMID 15519698.
- ^ Marfori M; Mynott A; Ellis JJ et al. (October 2010). “Molecular basis for specificity of nuclear import and prediction of nuclear localization”. Biochimica et Biophysica Acta 1813 (9): 1562–77. doi:10.1016/j.bbamcr.2010.10.013. PMID 20977914.
- ^ “A conserved mRNA export machinery coupled to pre-mRNA splicing”. Cell 108 (4): 523–31. (February 2002). doi:10.1016/S0092-8674(02)00627-X. PMID 11909523.
- ^ a b 能行, 松浦「核-細胞質間高分子輸送の構造生物学」『生物物理』第51巻第5号、日本生物物理学会、2011年、208–213頁、doi:10.2142/biophys.51.208、ISSN 0582-4052。
- ^ Lange, Allison; Mills, Ryan E.; Lange, Christopher J.; Stewart, Murray; Devine, Scott E.; Corbett, Anita H. (2007-02-23). “Classical nuclear localization signals: definition, function, and interaction with importin alpha”. The Journal of Biological Chemistry 282 (8): 5101–5105. doi:10.1074/jbc.R600026200. ISSN 0021-9258. PMC 4502416. PMID 17170104.
- ^ Stewart, Murray (2007-3). “Molecular mechanism of the nuclear protein import cycle”. Nature Reviews. Molecular Cell Biology 8 (3): 195–208. doi:10.1038/nrm2114. ISSN 1471-0072. PMID 17287812.
- ^ Hutten, Saskia; Kehlenbach, Ralph H. (2007-4). “CRM1-mediated nuclear export: to the pore and beyond”. Trends in Cell Biology 17 (4): 193–201. doi:10.1016/j.tcb.2007.02.003. ISSN 1879-3088. PMID 17317185.
- ^ Okamura, Masumi; Inose, Haruko; Masuda, Seiji (2015-03-20). “RNA Export through the NPC in Eukaryotes”. Genes 6 (1): 124–149. doi:10.3390/genes6010124. ISSN 2073-4425. PMC 4377836. PMID 25802992.
- ^ Iglesias, Nahid; Stutz, Françoise (2008-06-18). “Regulation of mRNP dynamics along the export pathway”. FEBS letters 582 (14): 1987–1996. doi:10.1016/j.febslet.2008.03.038. ISSN 0014-5793. PMID 18394429.
- ^ Cenik, C (2011). “Genome analysis reveals interplay between 5' UTR introns and nuclear mRNA export for secretory and mitochondrial genes.”. PLoS Genetics 7 (4): e1001366. doi:10.1371/journal.pgen.1001366. PMC 3077370. PMID 21533221.
- ^ a b “Dynamics of nuclear pore complex organization through the cell cycle”. Current Opinion in Cell Biology 16 (3): 314–21. (June 2004). doi:10.1016/j.ceb.2004.04.001. PMID 15145357.
- ^ Rabut, Gwénaël; Lénárt, Péter; Ellenberg, Jan (2004-6). “Dynamics of nuclear pore complex organization through the cell cycle”. Current Opinion in Cell Biology 16 (3): 314–321. doi:10.1016/j.ceb.2004.04.001. ISSN 0955-0674. PMID 15145357.
- ^ Walther, Tobias C.; Alves, Annabelle; Pickersgill, Helen; Loïodice, Isabelle; Hetzer, Martin; Galy, Vincent; Hülsmann, Bastian B.; Köcher, Thomas et al. (2003-04-18). “The conserved Nup107-160 complex is critical for nuclear pore complex assembly”. Cell 113 (2): 195–206. ISSN 0092-8674. PMID 12705868.
- ^ “Steps in the assembly of replication-competent nuclei in a cell-free system from Xenopus eggs”. The Journal of Cell Biology 106 (1): 1–12. (January 1988). doi:10.1083/jcb.106.1.1. PMC 2114961. PMID 3339085.
- ^ “Steps of nuclear pore complex disassembly and reassembly during mitosis in early Drosophila embryos”. Journal of Cell Science 114 (Pt 20): 3607–18. (October 2001). PMID 11707513.
- ^ Güttinger, Stephan; Laurell, Eva; Kutay, Ulrike (2009-3). “Orchestrating nuclear envelope disassembly and reassembly during mitosis”. Nature Reviews. Molecular Cell Biology 10 (3): 178–191. doi:10.1038/nrm2641. ISSN 1471-0080. PMID 19234477.
- ^ Laurell, Eva; Beck, Katja; Krupina, Ksenia; Theerthagiri, Gandhi; Bodenmiller, Bernd; Horvath, Peter; Aebersold, Ruedi; Antonin, Wolfram et al. (2011-02-18). “Phosphorylation of Nup98 by multiple kinases is crucial for NPC disassembly during mitotic entry”. Cell 144 (4): 539–550. doi:10.1016/j.cell.2011.01.012. ISSN 1097-4172. PMID 21335236.
- ^ Markossian, Sarine; Suresh, Subbulakshmi; Osmani, Aysha H.; Osmani, Stephen A. (2015-02-15). “Nup2 requires a highly divergent partner, NupA, to fulfill functions at nuclear pore complexes and the mitotic chromatin region”. Molecular Biology of the Cell 26 (4): 605–621. doi:10.1091/mbc.E14-09-1359. ISSN 1059-1524. PMC 4325833. PMID 25540430.
- ^ De Souza, Colin P. C.; Osmani, Aysha H.; Hashmi, Shahr B.; Osmani, Stephen A. (2004). “Partial Nuclear Pore Complex Disassembly during Closed Mitosis in Aspergillus nidulans”. Current Biology 14 (22): 1973–1984. doi:10.1016/j.cub.2004.10.050. ISSN 0960-9822. PMID 15556859.
- ^ Souza, Colin P. C. De; Osmani, Stephen A. (2007-09-01). “Mitosis, Not Just Open or Closed”. Eukaryotic Cell 6 (9): 1521–1527. doi:10.1128/EC.00178-07. ISSN 1535-9778. PMC 2043359. PMID 17660363.
- ^ Asakawa, Haruhiko; Kojidani, Tomoko; Mori, Chie; Osakada, Hiroko; Sato, Mamiko; Ding, Da-Qiao; Hiraoka, Yasushi; Haraguchi, Tokuko (2010-11-09). “Virtual breakdown of the nuclear envelope in fission yeast meiosis”. Current biology: CB 20 (21): 1919–1925. doi:10.1016/j.cub.2010.09.070. ISSN 1879-0445. PMID 20970342.
外部リンク 編集
- Histology image: 20104loa — ボストン大学の組織学学習システム
- Nuclear pore - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス(英語)
- Nuclear Pore Complex animations
- Nuclear Pore Complex illustrations
- 3D electron microscopy structures of the NPC and constituent proteins from the EM Data Bank(EMDB)
- NCDIR - National Center for the Dynamic Interactome