アポトソーム
アポトソーム(英: apoptosome)は、アポトーシスの過程で形成される巨大なタンパク質構造体である。アポトソームの形成は、内因性・外因性の細胞死刺激に応答してミトコンドリアから放出されたシトクロムcによって開始される。こうした細胞死をもたらす刺激には、DNA損傷やウイルス感染から、オタマジャクシの尾の消失をもたらす発生過程の指示まで多様な種類がある。
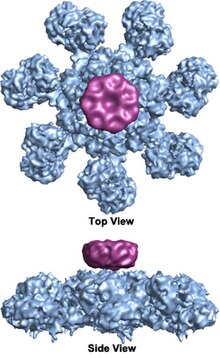
哺乳類細胞では、放出されたシトクロムcは細胞質基質のApaf-1タンパク質に結合し、アポトソームの形成を促進する。初期の生化学的研究ではシトクロムcとApaf-1が2:1で結合してアポトソームが形成されることが示唆されていたが、近年の構造解析ではシトクロムcとApaf-1の量比は1:1であることが示唆されている。3つ目の構成要素としてヌクレオチドdATPがApaf-1に結合することが示されているが、その正確な役割に関してはいまだ議論がある。哺乳類のアポトソームの結晶は得られていないが、2002年にヒトApaf-1/シトクロムcアポトソームがcryo-EMによって低分解能(20 Å)で可視化され、7回対称軸を有する車輪型構造が明らかにされた。ヒトのアポトソームの構造は中程度の分解能(9.5 Å)で解かれており、複合体中のApaf-1の各ドメイン(CARD、NB/ARC、WD40)とシトクロムcの位置が一意に特定されている。また、不活性型Apaf-1サブユニット単量体の結晶構造も得られている(PDB: 3SFZ)[1][2]。
形成されたアポトソームは、カスパーゼ-9前駆体(プロカスパーゼ-9)をリクルートして活性化する。活性化されたカスパーゼ-9は、イニシエーターカスパーゼとしてエフェクターカスパーゼを活性化し、アポトーシスにつながるカスケードを開始する。
歴史 編集
アポトソーム(apoptosome)という用語は、辻本賀英による1998年の論文"Role of Bcl-2 family proteins in apoptosis: apoptosomes or mitochondria?"[3]で初めて導入された。一方、この用語が用いられるようになる以前から、アポトソームは"ternary complex"(三者複合体)として認識されていた。この複合体にはカスパーゼ-9とBcl-xLが関係しており、それぞれApaf-1の特定のドメインに結合するとされ、この複合体の形成は哺乳類の細胞死を調節する役割を果たすと考えられていた[4]。そして1998年12月に、Apaf-1はプロカスパーゼ-9の活性化を介してアポトーシスを調節する因子であることを示す論文が発表された[5]。
アポトソームの定義となる基準は1999年に示された。まず巨大な複合体(1.3 MDa以上)であること、そしてその形成に際してATPまたはdATPの高エネルギー結合の加水分解を必要とすること、最後にその機能的な形態においてプロカスパーゼ-9を活性化する能力を持つことである。この複合体の形成はアポトーシス過程の回帰不能点となる。Apaf-1とシトクロムからなる安定な多量体タンパク質複合体はこの基準に合致し、現在ではこの複合体がアポトソームと呼ばれている[6]。アポトソームは複数のプロカスパーゼ-9分子を近接させて互いに切断を行わせる必要があること、そしてシトクロムcの非特異的漏出によってアポトーシスが生じることがないよう活性化の閾値を高くする必要がある、という2つの理由により多量体タンパク質複合体となっていると考えられた[6]。
アポトソームがプロカスパーゼ-9の活性化因子であることが確立されると、ヒト白血病細胞、卵巣がん、ウイルス感染による不死化などにおいて、この経路の変異が重要な研究領域となった[7][8][9]。また、Apaf-1を欠損した細胞により、Apaf-1、すなわちアポトソームに非依存的な細胞死経路も存在することが示された。この経路はカスパーゼ-3やカスパーゼ-9にも依存していない[10]。
構造 編集
アポトソームはアダプタータンパク質Apaf-1を中心として組み立てられる、複数の分子からなるホロ酵素複合体である。ミトコンドリアを介したアポトーシス経路によって組み立てられ、組み立てにはいくつかの種類のストレスシグナルによる刺激が必要である。
アポトソームが形成されるためには、ATP/dATPとシトクロムcが細胞質基質に存在することが必要である[11]。ストレス刺激によってシトクロムcの細胞質への放出が開始され、シトクロムcはApaf-1のC末端に位置する、複数のWD40リピートからなる領域に結合する[12]。Apaf-1のオリゴマー化は、N末端のCARDモチーフへのプロカスパーゼ-9のリクルートを伴って生じるようである[12]。アポトソームは、アポトーシスの内因性経路におけるカスパーゼの活性化を開始する[11]。
7回対称軸を有するアポトソームの車輪型七量体複合体構造はクライオ電顕によって27 Åの分解能で初めて明らかにされ、この複合体のサイズは約1 MDaと計算された[12]。この車輪型粒子には、7個のスポークからなる中心部のハブが存在する。スポークの遠位部にはY字型構造が存在する[11]。ハブドメインは曲がったアームによって、このYドメインと連結されている。各Yドメインは大小2つのローブから構成され、シトクロムcの結合部位は2つのローブの間に位置する[11]。このアポトソーム構造の分解能は比較的低かったため、アポトソームの構成に関して2つの対立するモデルが提唱された。1つのモデルでは、NODドメイン(nucleotide-binding and oligomerization domain)が中心部のハブを形成し、CARDドメインはその上部で比較的遊離した形でリングを形成するとされた[12]。もう一方のモデルでは、Apaf-1は伸長した形で複合体を形成しており、N末端のCARDとヌクレオチド結合領域が中心部のハブを形成し、13個のWD40リピートが2つのローブを構成する。そして大きなローブは7個のリピート、小さなローブは6個のリピートから構成される。各カスパーゼ-9分子は中心部のハブに位置するCARDドメインに結合し、ドーム型構造を形成するとされた[11]。この論争は、ヒトアポトソーム/プロカスパーゼ-9 CARD複合体の高分解能構造によって解決された[1]。アポトソームがプロカスパーゼ-9を結合すると、Apaf-1のCARDとプロカスパーゼ-9のCARDはアポトソームのプラットフォーム構造の上に柔軟なディスク様構造を形成する[1]。WD40リピートの数も13個ではなく15個であり、7枚のブレードからなるβプロペラと8枚のブレードからなるβプロペラを構成することが示された[1]。
複合体内のプロカスパーゼ-9とApaf-1の量比はおよそ1:1であることが生化学的解析から示され[6]、さらに定量的な質量分析によって確証された[13]。また、Apaf-1とシトクロムcの量比も1:1であることが示された[1]。アポトソームへのシトクロムcの安定的な取り込みはApaf-1のオリゴマー化後である必要があるかどうかに関しては多少の議論があるが、近年の構造データはシトクロムcがヒトアポトソームのオリゴマー化状態を安定化するという考えを支持している[1]。しかしながら、線虫やショウジョウバエといった哺乳類以外の生物種では、アポトソームの組み立てにシトクロムcは必要ではない可能性がある[14]。また、他の分子、最も特筆すべきものとしてはカスパーゼ-3がアポトソームと共精製されることが報告されており[6]、アポトソーム/プロカスパーゼ-9複合体に結合可能であることが示されている[13]。
Apaf-1はアポトソームの骨格を形成する。Apaf-1には、N末端のカスパーゼリクルートドメイン(CARD、1–90番残基)、中心部のヌクレオチド結合・オリゴマー化領域(NB-ARC/NOD、128–586番残基)、C末端のWD40領域(613–1248番残基)という主に3つの領域が存在し、各単量体のサイズは約140 kDaである[12]。
N末端のCARDドメインには短いリンカーとヌクレオチド結合ドメイン(NBD)が続く。NBDには保存されたWalkerボックスA(Pループ、155–161番残基)とB(239–243番残基)が含まれている[12]。WalkerボックスA/BはdATP/ATPとMg2+の結合に重要である[1][12]。NBDの後には小さなヘリカルドメイン(HD1)、2つ目のリンカー、そして保存されたウィングドヘリックスドメイン(WHD)が続く[12]。NBD、HD1、WHDはNOD領域を構成し、AAA+ファミリーに属するATPアーゼドメインを構成する[1][12]。NOD領域とWD40領域の間にはスーパーヘリカルドメイン(HD2)が存在する[1]。WD40領域はそれぞれ8つと7つのブレードからなる2つのβプロペラを形成し、両者の間はリンカーで連結されている[1]。
その他の生物種のアポトソーム 編集
これまでの記載は、ヒトのアポトソームに関するものである。他の生物種のアポトソーム複合体構造は類似点が多く存在する一方で、そのサイズやサブユニット数は大きく異なる。Apaf-1のショウジョウバエホモログはDarkと呼ばれ、8つのサブユニットからなるリング構造を形成する(PDB: 4V4L)[14]。線虫ホモログはCED-4と呼ばれ、これも八量体であるがそのサイズはかなり小さく(PDB: 3LQQ)、シトクロムcを結合する領域が含まれていない[15]。
作用機序 編集
開始 編集
アポトソームの作用の開始段階は、プログラム細胞死経路の第一段階に対応する。動物におけるアポトーシスは、膜貫通受容体に細胞外リガンドが結合することで開始される外因性経路と、ミトコンドリアで生じる内因性経路が存在する[16]。内因性経路はミトコンドリアからのシトクロムcの放出を伴い、放出されたシトクロムcは細胞質基質に位置するApaf-1に結合する[16][17]。このように、アポトソームの作用の開始にはシトクロムcの放出が必要である。放出はいくつかの方法で調節されており、最も重要なものはカルシウムイオン濃度の検知によるものである[16]。
シトクロムcの放出 編集
シトクロムcの放出は2通りの方法で行われていることが提唱されている。1つ目の機構では、ミトコンドリアが細胞死誘導シグナルを受け取った際に、ミトコンドリア膜透過性遷移孔(PTP)が形成され、膜間腔のタンパク質が放出される。PTPは電位依存性アニオンチャネル(VDAC)、内膜タンパク質のアデニンヌクレオチド輸送体(AdNT)、マトリックスタンパク質のシクロフィリンD(CyD)から構成される。この孔はミトコンドリアの膨潤や、外膜の破裂をもたらす。こうした透過性の変化によって、シトクロムcなどのタンパク質が細胞質基質へ放出される。こうした変化はミトコンドリア透過性遷移(MPT)の原因となっている可能性が高く、ミトコンドリア膜電位の消失やATP産生の停止が生じる。シトクロムc放出の2つ目の機構はPTP非依存的なものであり、VDACのみが関与する。Bcl-2ファミリーのアポトーシス促進タンパク質はVDACの開口を誘導する。その結果、同様に膜間腔タンパク質の放出が引き起こされ、シトクロムcの放出、そしてMPTが生じる[17]。
Apaf-1 編集
シトクロムc非存在下 編集
シトクロムcが存在しない場合、Apaf-1は単量体として存在する。WD40ドメインは折り返され、Apaf-1は自己阻害状態に維持されていると考えられている。さらに、いくつかの領域が強固に結合しており、Apaf-1は他の因子を結合することができない状態となっている。質量分析により、この「ロックされた」自己阻害状態では、Apaf-1のATPアーゼドメインにはADPが結合していることが明らかにされている[16]。この状態ではApaf-1は単量体であり、カスパーゼを活性化する能力を持たない。
シトクロムc存在下 編集
シトクロムcはApaf-1のWD40ドメインに結合する。その結果「ロック」は解除される[1][16]。しかしながら、CARDとNB-ARCは自己阻害状態のままである[16]。ATPアーゼドメインにADPが存在している場合にはオリゴマー化は阻害されている。CARDドメインの自己阻害はApaf-1にdATP/ATPが結合した際に解除され、七角形構造へのオリゴマー化を可能にとなってCARDドメインはカスパーゼ-9への結合が可能となる[1][6][16]。ATPアーゼドメイン内に変異を導入することでタンパク質は不活性化されるが、ADP-ATP交換を制御している機構は不明確である[1][6]。このオリゴマー化は7分子のシトクロムc、7分子のApaf-1、そして十分量のdATP/ATPが存在している場合のみに生じる[6]。Apaf-1のATPアーゼドメインはAAA+ファミリーに属し、このファミリーは他のATPアーゼドメインを連結して六量体または七量体を形成することが知られている[16]。アポトソームは、NB/ARCドメインが中心に位置するような形で7つのApaf-1分子が車輪型に配置された際に、活性化状態とみなされる[1][16]。
活性型アポトソームの作用 編集
この機能的アポトソームは、カスパーゼ-9活性化のためのプラットフォームとなる[1][16]。カスパーゼ-9は酵素前駆体の形で細胞質基質に存在し、細胞内には約20 nMの濃度で存在していると考えられている[16]。酵素前駆体が活性型となるためには切断は絶対的に必要であるわけではないが[16]、切断を受けることでその活性は大幅に上昇する[13]。アポトソームで生じるカスパーゼ-9の切断機構に関しては、2分子のカスパーゼ-9が切断前に二量体化するための場所をアポトソームが提供しているという仮説や、アポトソーム上でのカスパーゼ-9の切断は単量体のままで起こるという仮説が提唱されている[13][16]。いずれにせよ、アポトソーム上で生じたカスパーゼ-9の活性化はカスパーゼカスケード全体の活性化、そして細胞死をもたらす。カスパーゼカスケードの活性化に多量体タンパク質複合体が必要とされる進化的な理由としては、微量のシトクロムcによってアポトソームが偶発的に引き起こされることがないよう保証するためであることが示唆されている[6]。
臨床との関係 編集
アポトソームの変異 編集
アポトーシスは正常な体の機能に必要であり、アポトーシス経路の変異は体に破滅的な影響をもたらす。アポトーシス経路の変異が原因となる疾患は多く、アポトソーム活性の欠損はがん、そして過剰なアポトソーム活性はアルツハイマー病の原因となるほか、パーキンソン病やハンチントン病など他の多くの神経変性疾患とも関係している[18]。アルツハイマー病、パーキンソン病、ハンチントン病といった神経変性疾患は全て加齢と関連した疾患であり、アポトーシスの増大によって、まだ機能し、組織の機能に寄与することができる細胞が死滅する現象が関係している。Apaf-1-ALTは前立腺がんにみられる変異型Apaf-1であり、339-1248番残基を欠損している。Apaf-1-ALTはアポトソームの組み立てに重要な構造的構成要素を失っており、アポトソームを形成できないことが構造研究から示されている[1]。
がん治療におけるアポトソームの標的化 編集
アポトーシスの阻害はがんの重要な特徴の1つであり、この阻害を操作・克服する方法を見つけることが新たながん治療法開発において重要となっている[19]。アポトソームの場合、外部刺激によってアポトソームを活性化することでアポトーシスが生じさせ、変異細胞を除去することができると考えられる。特定のアポトーシス調節因子を標的として過剰な細胞死や細胞死の欠如といった異常を是正するため、組換え生体分子、アンチセンスオリゴ、遺伝子治療や古典的なコンビナトリアルケミストリーなど多方面からのアプローチが行われている[18]。
Apaf-1/カスパーゼ-9アポトソームの形成はアポトーシスカスケードにおける重要なイベントであり、活性型アポトソームの形成を阻害または安定化する薬剤候補の同定は、過剰または不十分なアポトーシスによって特徴づけられる疾患の治療へ向けた最適な戦略となる[18]。タウリンは、ミトコンドリアの機能不全に影響を及ぼすことなく、Apaf-1/カスパーゼ-9型アポトソームの形成を阻害することで、心筋細胞における虚血誘発性のアポトーシスを阻害することが発見されている。タウリンによるアポトソーム形成阻害の推定機構は、アポトソームの基本的構成要素であるカスパーゼ-9の発現低下によるものである。一方、Apaf-1やカスパーゼ-9にはアポトソーム非依存的な役割もあるため、こうした因子濃度の変化は他の機能にも影響を及ぼす可能性がある。このように、有望な実験的データが得られた場合でも、いくつかの未解決な問題によって臨床使用に制限が生じる場合がある[18]。そのため特に重要となるのは、アポトソームの構成要素の転写レベルを変化させることなく、細胞内のタンパク質間相互作用を標的とすることで、アポトソームの安定性や活性を阻害する化合物である[18]。近年のアポトソームに関する構造研究の進展は、アポトソーム複合体を標的とした治療デザインにおいて有用なツールとなる可能性がある[1][13]。
出典 編集
- ^ a b c d e f g h i j k l m n o p q r s t Yuan, Shujun; Yu, Xinchao; Topf, Maya; Ludtke, Steven J.; Wang, Xiaodong; Akey, Christopher W. (2010-05-12). “Structure of an apoptosome-procaspase-9 CARD complex”. Structure (London, England: 1993) 18 (5): 571–583. doi:10.1016/j.str.2010.04.001. ISSN 1878-4186. PMC 2874686. PMID 20462491.
- ^ T. F. Reubold; S. Wohlgemuth; S. Eschenburg (2011). “Crystal structure of full-length Apaf-1: how the death signal is relayed in the mitochondrial pathway of apoptosis”. Structure 19 (8): 1074–1083. doi:10.1016/j.str.2011.05.013. PMID 21827944.
- ^ “Role of Bcl-2 family proteins in apoptosis: apoptosomes or mitochondria?”. Genes to Cells 3 (11): 697–707. (November 1998). doi:10.1046/j.1365-2443.1998.00223.x. PMID 9990505.
- ^ Pan, G.; O'Rourke, K.; Dixit, V. M. (1998). “Caspase-9, Bcl-XL, and Apaf-1 Form a Ternary Complex”. Journal of Biological Chemistry 273 (10): 5841–845. doi:10.1074/jbc.273.10.5841. PMID 9488720.
- ^ Hu, Ding; Spencer; Nunez (1998). “WD-40 Repeat Region Regulates Apaf-1 Self-association and Procaspase-9 Activation”. Journal of Biological Chemistry 273 (50): 33489–3494. doi:10.1074/jbc.273.50.33489. PMID 9837928.
- ^ a b c d e f g h “An APAF-1.cytochrome c multimeric complex is a functional apoptosome that activates procaspase-9”. The Journal of Biological Chemistry 274 (17): 11549–56. (April 1999). doi:10.1074/jbc.274.17.11549. PMID 10206961.
- ^ “Apaf-1 protein deficiency confers resistance to cytochrome c-dependent apoptosis in human leukemic cells”. Blood 98 (2): 414–21. (July 2001). doi:10.1182/blood.v98.2.414. PMID 11435311.
- ^ “Caspase-8 and Apaf-1-independent caspase-9 activation in Sendai virus-infected cells”. The Journal of Biological Chemistry 277 (33): 29817–9824. (August 2002). doi:10.1074/jbc.M111898200. PMID 12021264.
- ^ “Defective cytochrome c-dependent caspase activation in ovarian cancer cell lines due to diminished or absent apoptotic protease activating factor-1 activity”. The Journal of Biological Chemistry 276 (36): 34244–51. (September 2001). doi:10.1074/jbc.M011778200. PMID 11429402.
- ^ “Apoptosome-independent pathway for apoptosis. Biochemical analysis of APAF-1 defects and biological outcomes”. The Journal of Biological Chemistry 278 (32): 29571–80. (August 2003). doi:10.1074/jbc.M302924200. PMID 12773531.
- ^ a b c d e “Apoptosome Structure and Regulation”. Apoptosome: 27–39. (2010). doi:10.1007/978-90-481-3415-1_2. ISBN 978-90-481-3414-4.
- ^ a b c d e f g h i j k l “Three-dimensional structure of the apoptosome: implications for assembly, procaspase-9 binding, and activation”. Molecular Cell 9 (2): 423–32. (February 2002). doi:10.1016/s1097-2765(02)00442-2. PMID 11864614.
- ^ a b c d e Yuan, Shujun; Yu, Xinchao; Asara, John M.; Heuser, John E.; Ludtke, Steven J.; Akey, Christopher W. (2011-08-10). “The holo-apoptosome: activation of procaspase-9 and interactions with caspase-3”. Structure (London, England: 1993) 19 (8): 1084–1096. doi:10.1016/j.str.2011.07.001. ISSN 1878-4186. PMC 3155825. PMID 21827945.
- ^ a b Yuan, Shujun; Yu, Xinchao; Topf, Maya; Dorstyn, Loretta; Kumar, Sharad; Ludtke, Steven J.; Akey, Christopher W. (2011-01-12). “Structure of the Drosophila apoptosome at 6.9 å resolution”. Structure (London, England: 1993) 19 (1): 128–140. doi:10.1016/j.str.2010.10.009. ISSN 1878-4186. PMC 3053581. PMID 21220123.
- ^ S. Qi; Y. Pang; Q. Hu; Q. Liu; H. Li; Y. Zhou; T. He; Q. Liang et al. (2010). “Crystal structure of the Caenorhabditis elegans apoptosome reveals an octameric assembly of CED-4”. Cell 141 (3): 446–457. doi:10.1016/j.cell.2010.03.017. PMID 20434985.
- ^ a b c d e f g h i j k l m “The apoptosome: signalling platform of cell death”. Nature Reviews. Molecular Cell Biology 8 (5): 405–13. (May 2007). doi:10.1038/nrm2153. PMID 17377525.
- ^ a b Diamond, Mark; McCabe, Paul F. (2007-04-06), Logan, David C., ed. (英語), The Mitochondrion and Plant Programmed Cell Death, John Wiley & Sons, Inc., pp. 308–334, doi:10.1002/9780470986592.ch10, ISBN 978-0-470-98659-2 2024年2月12日閲覧。
- ^ a b c d e “The apoptosome: emerging insights and new potential targets for drug design”. Pharmaceutical Research 25 (4): 740–51. (April 2008). doi:10.1007/s11095-007-9396-z. PMC 2279152. PMID 17674158.
- ^ “Cutting-edge apoptosis-based therapeutics: a panacea for cancer?”. BioDrugs 21 (5): 273–97. (2007). doi:10.2165/00063030-200721050-00001. PMID 17896835.