カナマイシンキナーゼ
カナマイシンキナーゼ(kanamycin kinase、EC 2.7.1.95)は、以下の化学反応を触媒する酵素である。
- ATP + カナマイシン ADP + カナマイシン-3'-リン酸
従って、この酵素の基質はATPとカナマイシンの2つ、生成物はADPとカナマイシン-3'-リン酸の2つである。 この酵素は転移酵素、特にアルコールを受容体とするホスホトランスフェラーゼに分類される。この酵素の系統名は、ATP:カナマイシン 3'-O-ホスホトランスフェラーゼ(ATP:kanamycin 3'-O-phosphotransferase)である。ネオマイシン-カナマイシンホスホトランスフェラーゼ(neomycin-kanamycin phosphotransferase)とも呼ばれる[2]。
この酵素はカナマイシンだけでなく、4,6-二置換アミノグリコシドの3'-ヒドロキシル基に対してリン酸基の付加を触媒するため、アミノグリコシド-3'-ホスホトランスフェラーゼ(aminoglycoside-3'-phosphotransferase、APH(3'))またはアミノグリコシドキナーゼ(aminoglycoside kinase)とも呼ばれる。APH(3')はさらに、3'-ヒドロキシル基が存在しない4,5-二置換アミノグリコシドに対しては5'-ヒドロキシル基のリン酸化を行い、3'-ヒドロキシル基、5'-ヒドロキシル基の双方を持つアミノグリコシドに対してはその双方のリン酸化を行う[3][4]。アミノグリコシドは生物学的条件下で主に正に帯電しているため、負に帯電した核酸の主鎖に結合してタンパク質合成を阻害し、細菌の細胞成長を効果的に阻害する[5]。APH(3')はアミノグリコシドにリン酸基を導入し、立体障害と静電的反発によって結合親和性を低下させることでアミノグリコシドの作用を効果的に阻害する[6]。 APH(3')は、主に特定種のグラム陽性菌に存在する[7][8][9]。

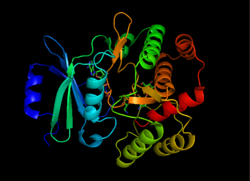
構造
編集APH(3')は熱力学的に二量体型を好み、2つの同一なAPH(3')単量体はCys19とCys156の2つのジスルフィド結合で連結され、活性部位は互いに向かい合っている[3][10]。しかし各単量体の活性部位は大きく離れており、これらは協働的ではなく互いに独立して機能していることが示唆される。さらに、APH(3')の二量体化は酵素活性に影響を与えない[3][10][11]。
各単量体はβシートに富むN末端ローブとαヘリックスに富むC末端ローブの2つのローブから構成され、両者は12アミノ酸の領域で連結されている。N末端ローブは5本の逆平行βシートからなり、シート3と4の間にはαヘリックスが存在する。C末端ローブは中心となるコア領域(2本のαヘリックス、ヘアピンループとそれに続く4本のβシート)、挿入領域(ループで連結された2本のαヘリックス)、C末端領域(2本のαヘリックス)へと分割される[11]。2つのローブで覆われたポケットが酵素の活性部位を構成している[3]。ポケットの大部分は負に帯電したアミノ酸残基によって構成され、活性部位内で基質の正電荷を安定化し配向を整えている。さらに、このポケットは基質の曖昧性にも寄与していると考えられており、いくつかの異なる種類のアミノグリコシドが結合し安定化される[7]。
機構
編集APH(3')に関する初期の研究は3'-ヒドロキシル基によるγ-リン酸の求核攻撃機構を支持していたが、より近年の研究は、APH(3')は解離機構によるATPからアミノグリコシドへのγ-リン酸の転移を触媒することを示唆している。リン酸の転移には基質の脱プロトン化ではなく、メタリン酸遷移状態の安定化が重要である[9][12]。さらに、APH(3')にはATP結合後に酵素の活性部位を閉じるNPL(nucleotide positioning loop)と呼ばれる領域が存在し、この領域によって3'-ヒドロキシル基のリン酸化が促進される。中でもリン酸基の正確な配置に重要なのは、Ser27とMet26である。まず、Asn195とAsp208によって安定化された2つのマグネシウムイオンがATPの活性部位への結合を促進し、β-、γ-リン酸を正しい向きに配置する。その後NPLのコンフォメーションが変化し、Ser27とβ-リン酸の間で水素結合が形成される。基質の結合に伴ってAPH(3')にはさらなるコンフォメーション変化が生じ、Ser27の主鎖のアミドがβ-リン酸とγ-リン酸の整列を破壊し、γ-リン酸結合を弱める。Met26の主鎖のアミドはメタリン酸と水素結合を形成して遷移状態を安定化し、マグネシウムイオン(下図のMg1)がγ-リン酸結合を長くして壊すことで、ヒドロキシル基は効率的にリン酸化される[9]。
進化と生物学的機能
編集APH(3’)中心部のコア領域には、真核生物に存在するセリン/スレオニン型やチロシン型のプロテインキナーゼのコンフォメーションと高度の類似性が存在する。さらに、X線結晶構造解析と活性部位の主要残基の変異体解析からもAPH(3’)と真核生物のプロテインキナーゼの関連性は支持されるが、一次構造上の配列保存性はほとんどみられない[3][9][11]。かつてはセリン/スレオニン/チロシンキナーゼは真核生物にのみ存在すると考えられていたが、原核生物にも存在することがいくつかの研究から示唆されている[13][14]。さらに、アミノグリコシドの生合成は特定の段階でヒドロシル基のリン酸化を必要とすることが知られている。そのため、APH(3’)と他のプロテインキナーゼはアミノグリコシドの代謝経路の酵素に起源を持ち、こうした抗生物質の宿主細菌細胞に対する毒性を中和するために発達したと推測されている[11][15]。
研究における利用
編集遺伝子工学において、アミノグリコシド耐性遺伝子は正しく形質転換された細菌を選択する目的で広く利用されている。研究対象の遺伝子を効果的に発現するためには、ベクターとなるプラスミドを構築する際に抗生物質耐性を組み込むことが重要である。アミノグリコシド(カナマイシンやネオマイシン)などの抗生物質は、プラスミドを取り込まなかった細胞の選択的破壊を目的として増殖期に培地に添加される。
出典
編集- ^ Fong, DH, Berghuis, AM (2002). Crystal Structure Of 3',5"-Aminoglycoside Phosphotransferase Type IIIa ADP Kanamycin A Complex. doi:10.2210/pdb1l8t/pdb.
- ^ “Catalytic mechanism of enterococcal kanamycin kinase (APH(3')-IIIa): viscosity, thio, and solvent isotope effects support a Theorell-Chance mechanism”. Biochemistry 35 (26): 8680–5. (1996). doi:10.1021/bi9603884. PMID 8679630.
- ^ a b c d e f Wright, GD, Thompson, PR (1999). “Aminoglycoside phosphotransferases: proteins, structure, and mechanism.”. Front Biosci 4 (1–3): D9–21. doi:10.2741/wright. PMID 9872733.
- ^ Thompson, PR, Hughes, DW, Wright, GD (1996). “Regiospecificity of aminoglycoside phosphotransferase from Enterococci and Staphylococci (APH(3')-IIIa).”. Biochemistry 35 (26): 8686–95. doi:10.1021/bi960389w. PMID 8679631.
- ^ Cavallo, G, Martinetto, P (1981). “The mechanism of action of aminoglycosides”. G Batteriol Virol Immunol 74 (7–12): 335–46. PMID 6182050.
- ^ Kotra, LP, Haddad J, Mobashery, S (2000). “Aminoglycosides: Perspectives on Mechanisms of Action and Resistance and Strategies to Counter Resistance”. Antimicrobial Agents and Chemotherapy 44 (12): 3249–56. doi:10.1128/aac.44.12.3249-3256.2000. PMC 90188. PMID 11083623.
- ^ a b Fong, DH, Berghuis, AM (2002). “Substrate promiscuity of an aminoglycoside antibiotic resistance enzyme via target mimicry”. The EMBO Journal 21 (10): 2323–31. doi:10.1093/emboj/21.10.2323. PMC 126009. PMID 12006485.
- ^ Gray, GS, Fitch WM (1983). “Evolution of antibiotic resistance genes: the DNA sequence of a kanamycin resistance gene from Staphylococcus aureus.”. Mol Biol Evol 1 (1): 57–66. doi:10.1093/oxfordjournals.molbev.a040298. PMID 6100986.
- ^ a b c d e Thompson, PR, Boehr, DD, Berghuis, AM, Wright, GD (2002). “Mechanism of Aminoglycoside Antibiotic Kinase APH(3')-IIIa: Role of the Nucleotide Positioning Loop”. Biochemistry 41 (22): 7001–7. doi:10.1021/bi0256680. PMID 12033933.
- ^ a b McKay, GA, Thompson, PR, Wright, GD (1994). “Broad spectrum aminoglycoside phosphotransferase type III from Enterococcus: overexpression, purification, and substrate specificity.”. Biochemistry 33 (22): 6936–44. doi:10.1021/bi00188a024. PMID 8204627.
- ^ a b c d Hon, WC, McKay, GA, Thompson, PR, Sweet, RM, Yang, DSC, Wright, GD, Berhuis, AM (1997). “Structure of an Enzyme Required for Aminoglycoside Antibiotic Resistance Reveals Homology to Eukaryotic Protein Kinases.”. Cell 89 (6): 887–95. doi:10.1016/s0092-8674(00)80274-3. PMID 9200607.
- ^ Boehr, DD, Thompson, PR, Wright, GD (2001). “Molecular mechanism of aminoglycoside antibiotic kinase APH(3')-IIIa: roles of conserved active site residues.”. J Biol Chem 276 (26): 23929–36. doi:10.1074/jbc.m100540200. PMID 11279088.
- ^ Kennelly, PJ (1996). “Fancy meeting you here! A fresh look at "prokaryotic" protein phosphorylation.”. J Bacteriol 178 (16): 4759–64. doi:10.1128/jb.178.16.4759-4764.1996. PMC 178254. PMID 8759835.
- ^ Zhang, CC (1996). “Bacterial signalling involving eukaryotic-type protein kinases.”. Mol Microbiol 20 (1): 9–15. doi:10.1111/j.1365-2958.1996.tb02483.x. PMID 8861199.
- ^ Pierpersberg, W, Distler, J, Heinzel, P, Perez-Gonzalaez, JA (1988). “Antibiotic resistance by modification: Many resistance genes could be derived from cellular control genes in actinomycetes - a hypothesis”. Actinomycetologica 2 (2): 83–98. doi:10.3209/saj.2_83.
関連文献
編集- “Inactivation of kanamycin, neomycin, and streptomycin by enzymes obtained in cells of Pseudomonas aeruginoa”. Applied Microbiology 16 (9): 1276–81. (Sep 1968). PMC 547640. PMID 4970990.
- “The Streptococcus faecalis oxidases for reduced diphosphopyridine nucleotide. III. Isolation and properties of a flavin peroxidase for reduced diphosphopyridine nucleotide”. The Journal of Biological Chemistry 225 (1): 557–73. (Mar 1957). PMID 13416259.