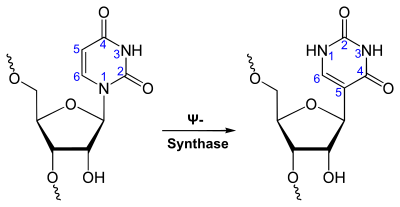シュードウリジン
シュードウリジン (英: pseudouridine、ギリシャ文字のΨや英文字のQで略記される)[1] は、ヌクレオシドであるウリジンの異性体。リボース環とウラシルの間のグリコシド結合のN-C結合がC-C結合に置き換わっている。RNAに知られている100を超える修飾塩基としては最も一般的で[2]、Ψはすべての生物種、ほとんどのRNAに見出されている[3][4]。
| シュードウリジン | |
|---|---|
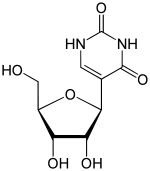
| |
5-[(2S,3R,4S,5R)-3,4-Dihydroxy-5-(hydroxymethyl)-oxolan-2-yl]-1H-pyrimidine-2,4-dione | |
5-(β-D-ribofuranosyl)pyrimidine-2,4(1H,3H)-dione | |
別称 psi-Uridine, 5-Ribosyluracil, beta-D-Pseudouridine, 5-(beta-D-Ribofuranosyl)uracil | |
| 識別情報 | |
| CAS登録番号 | 1445-07-4 |
| PubChem | 15047 |
| ChemSpider | 14319 |
| UNII | 7R0R6H6KEG |
| ChEBI | |
| |
| |
| 特性 | |
| 化学式 | C9H12N2O6 |
| モル質量 | 244.20 g/mol |
| 外観 | White granular powder |
| 水への溶解度 | Highly soluble in water. |
| 特記なき場合、データは常温 (25 °C)・常圧 (100 kPa) におけるものである。 | |
転写やその後の合成過程の後、RNAは化学的に異なる100以上の修飾を受ける。こうした修飾は転写後の段階でRNAの発現を調節している可能性があり、RNAの翻訳、局在、安定化などさまざまな役割を果たしている可能性がある。シュードウリジル化もそうした修飾の1つであり、ウリジンがC5-グリコシド結合型異性体であるシュードウリジンへ変換される。ウリジンには一般的なリボースのC1位とウラシルのN1位の間のC-N結合が存在するのに対し、シュードウリジンではリボースのC1位とウラシルのC5位がC-C結合を形成している。C-C結合は、より大きな回転自由度と立体配座の柔軟性を与える[3]。さらに、シュードウリジンにはN1位に余分な水素結合供与体が存在する。シュードウリジンは5-リボシルウラシルとしても知られ、tRNA、rRNA、snRNA、snoRNAなどの構造的RNAの構成要素として普遍的に存在している。近年では、タンパク質をコードするmRNAにも発見されている。シュードウリジンは最も豊富に存在する修飾塩基であり、生物の3つのドメインすべてに存在し、最初に発見された修飾塩基である。このヌクレオチドは「5番目のヌクレオチド」であると見なされており、酵母のtRNAの4%を占めている。この塩基修飾はRNAを安定化し、余分なイミノ基を介して水分子と水素結合を形成することで塩基のスタッキングを強化する。大腸菌Escherichia coliのrRNAにはシュードウリジンが11個存在し、酵母の細胞質のrRNAには30個、ミトコンドリアの21S rRNAには1つ、ヒトのrRNAには約100個存在する。これらからは、生物の複雑さとともにシュードウリジル化の程度が増大していることが示唆される。rRNAやtRNA中のシュードウリジンは局所的な構造を微調整して安定化し、mRNAの解読、リボソームの組み立てとプロセシング、そして翻訳に関する機能の維持を助けることが示されている[3][5][6]。snRNA中のシュードウリジンは、スプライソソームRNAとpre-mRNAとの相互作用を高め、スプライシングの調節を促進することが示されている[7]。
さまざまなRNAへの修飾と影響
編集tRNA
編集ΨはtRNAに普遍的に存在し、tRNAに共通した構造モチーフの形成を促進する。そうした構造モチーフの1つがTΨCステムループであり、Ψ55が関係している。ΨはDステムとアンチコドンステムループにも一般的にみられる。各構造モチーフにおいて、Ψの独特な物理化学的な性質は標準的なUでは不可能な構造安定化を可能にしている[3]。
翻訳時に、ΨはtRNA分子とrRNA、mRNAとの相互作用を調整する。Ψや他の修飾ヌクレオチドは、それらが存在するドメインの局所的構造に影響を与えるが、RNA分子全体のフォールディングに影響を与えることはない。アンチコドンステムループ(ASL)では、ΨはtRNAがリボソームに適切に結合するために重要であるようである。ΨはASLの動的な構造を安定化し、30Sリボソームへのより強固な結合を促進する。ASLのコンフォメーションの安定化は、翻訳時の適切なアンチコドン-コドン対合の維持を助ける。この安定性は、ペプチド結合の形成速度を低下させ、不正確なコドン-アンチコドン対合を排除するための時間を長くすることで、翻訳の正確性を向上させている可能性がある。しかしながら、tRNAのシュードウリジル化は細胞の生存に必須ではなく、アミノアシル化にも通常は必要とされない[3]。
mRNA
編集Ψは、タンパク質合成の鋳型となるmRNAにも存在している。mRNA中のΨ残基は終止コドンUAA、UGA、UAGのコーディング特性に影響を与える。これらの終止コドンのU→Ψへの修飾は、U→Cへの変異と同様に、ナンセンスサプレッションを促進する[8]。
rRNA
編集Ψは、全ドメインの生物とその細胞小器官のリボソームの大小のサブユニットに存在する。リボソームでは、Ψ残基はドメインII、IV、Vに密集しており、RNA-RNA間またはRNA-タンパク質間の相互作用を安定化している。Ψによってもたらされる安定性は、rRNAのフォールディングとリボソームの組み立てを補助している可能性がある。Ψは局所構造の安定性にも影響を与えている可能性があり、翻訳時のデコーディングの速度と正確性、校正機能に影響を与える[3]。
snRNA
編集Ψは、真核生物の主要なスプライセオソームのsnRNAにも存在している。snRNA中のΨ残基は多くの場合系統学的に保存されているが、分類群や生物種によって多少の差異が存在する。snRNA中のΨ残基は、通常RNA-RNA間またはRNA-タンパク質間の相互作用に関与する領域に位置しており、スプライセオソームの組み立てや機能に関与する。snRNA中のΨ残基は、スプライセオソームの適切なフォールディングや組み立て位に寄与し、pre-mRNAのプロセシングに必要不可欠である[3]。
シュードウリジンシンターゼ
編集シュードウリジル化は、転写後、すなわちRNAが形成された後に行われるRNA修飾である。この修飾を行うタンパク質はシュードウリジンシンターゼ(PUS)と呼ばれている。PUSは生物の全てのドメインに存在する。研究の大部分はPUSがtRNAを修飾する機構に関して行われており、そのためsnRNAやmRNAに関する機構は明確ではない。PUSタンパク質のRNAに対する特異性、構造、そして異性化機構はさまざまである。さまざまなPUSの構造は4つのファミリーに分類される。ファミリー内では、共通した活性配列と重要な構造モチーフが存在する[2]。
TruA
編集TruAドメインは、tRNA、snRNA、mRNAのさまざまな部位を修飾する。このファミリーによるウリジンの異性化機構にはまだ議論がある[6][9]。
PUS1は核内に存在し、tRNAのさまざまな部位、U2 snRNAU44、U6 snRNAのU28を修飾する。PUS1の発現は環境ストレス時に増加し、RNAスプライシングの調節に重要であることが示されている。また、PUS1は核内で合成されたtRNAを細胞質へ送る際にも必要である[6]。
PUS2はPUS1にきわめて類似しているが、ミトコンドリアに位置し、ミトコンドリアのtRNAのU27とU28のみを修飾する。ミトコンドリアのtRNAは他のtRNAと比較してシュードウリジンの量は少ない。他のミトコンドリア局在タンパク質と異なり、PUS2にはミトコンドリア標的化シグナルが見つかっていない[6]。
PUS3はPUS1と相同であるが、細胞質とミトコンドリアでtRNAのU38/39を修飾する。このタンパク質はTruAファミリーの中で最も保存性が高い。tRNAが不適切なフォールディングをしている際には、PUS3による修飾が低下する。tRNAの他にもノンコーディングRNAやmRNAを標的化するが、この修飾の重要性に関してはさらなる研究が必要である。ヒトでは、PUS3はPUS1とともに、SRA1ノンコーディングRNAを修飾する[6]。
TruB
編集TruBファミリーはミトコンドリアと核に位置するPUS4のみから構成される。PUS4は、tRNAのエルボーに位置する高度に保存されたU55を修飾する。ヒトのPUS4は、PUA(pseudouridine synthase and archaeosine trans-glycosylase)ドメインと呼ばれる結合ドメインを欠いている。PUS4はtRNAのTループ部分に対する配列特異性を有する。PUS4がmRNAを修飾する予備的なデータが存在するが、その確証にはさらなる研究が必要である。また、植物に感染するRNAウイルスであるブロムモザイクウイルスに対して特異的に結合する[6][10]。
TruD
編集TruDはさまざまなRNAの修飾を行うが、どのようにRNA基質が認識されているのかははっきりしない。PUS7はU2 snRNAの35位を修飾し、この修飾はヒートショックによって誘導される。他には細胞質のtRNAの13位とpre-tRNATyrの35位が修飾される。PUS7はmRNAのシュードウリジル化も行い、特異性はRNAの種類に依存しない。PUS7はUGUARの配列を認識し、2番目のUが修飾される。PUS7はヒートショック時に核から細胞質へ移行し、mRNAのシュードウリジル化を増加させる。修飾はRNAの核から細胞質への再局在と相関しており、ヒートショック時にmRNAの安定性を増加させていると考えられているが、さらなる研究が必要である[6][9]。
RluA
編集RluAドメインによる認識は、タンパク質の結合によって誘導される二次構造に対する形状相補性に依存しているようである[2]。
PUS5は研究は進んでいないが、ミトコンドリアに局在するシュードウリジンシンターゼである。PUS2と同様、ミトコンドリア標的化シグナル配列は見つかっていない。このタンパク質はミトコンドリアの21S rRNAのU2819を修飾する。PUS5はmRNA中の一部のウリジンを修飾する可能性があるが、これに関してもさらなるデータが必要である[6]。
PUS6は細胞質とミトコンドリアのtRNAのU31を修飾する酵素である。mRNAを修飾することも示されている[6]。
PUS8はRib2としても知られ、細胞質のtRNAのU32を修飾する。C末端にはDRAPデアミナーゼドメインが存在し、リボフラビンの生合成に関係している。RluAドメインとデアミナーゼドメインは完全に別個の機能であり、両者が互いに相互作用するかは不明である。PUS8は酵母では必須であるが、シュードウリジンの修飾ではなくリボフラビン合成と関係した機能のためであると考えられている[6]。
PUS9は、細胞質ではなくミトコンドリアのtRNAに対し、PUS8と同じ部位を修飾する酵素である。N末端にミトコンドリア標的化シグナルドメインが存在する唯一のPUSタンパク質である。PUS9はmRNAも修飾する可能性が示唆されており、基質特異性は低いと考えられる[6]。
シュードウリジンのシーケンシング技術
編集シュードウリジンは多数の異なる技術によって同定することができる。RNAやDNA中の修飾を同定する一般的な技術は液体クロマトグラフィー/質量分析(LC/MS)である。質量分析は分子を質量と電荷で分離する。ウリジンはとシュードウリジンは質量は同じであるが、電荷が異なる。液体クロマトグラフィーはカラム中での保持時間の差によって分離を行う[11]。シュードウリジンを同定する化学的手法では、CMC(N-cyclohexyl-N′-β-(4-methylmorpholinium)ethylcarbodiimide)と呼ばれる化合物が用いられる。CMCはシュードウリジンとウリジンの双方と結合するが、シュードウリジンのN3位に結合したCMCはアルカリ処理後も保持されるため、シュードウリジンを特異的にラベル化し、ウリジンから区別することができる[12]。シュードウリジンに結合したCMCは、シグナル分子でタグ付けを行ってイメージングに利用することもできる。この手法をハイスループット化する研究が行われている[13]。
医学との関係
編集シュードウリジンは、近傍の糖-リン酸主鎖にわずかではあるが重大な影響を与え、塩基の積み重なり (スタッキング) も強化する。こうした効果は、RNA中に存在するシュードウリジン残基のすべてではないが大部分で、生物学的な役割の基礎となっている可能性がある。tRNAやrRNA中の特定のシュードウリジン残基を喪失する遺伝的変異によって、翻訳に困難が生じ、生育速度は低下し、野生株との混合培養では野生株に競合することができなくなる。シュードウリジン修飾はMLASA(乳酸アシドーシスと鉄芽球性貧血を伴うミトコンドリアミオパチー)や先天性角化異常症などのヒトの疾患への関与も示唆されている[6]。先天性角化異常症とHoyeraal-Hreidarsson症候群は、シュードウリジンシンターゼであるジスケリンをコードするDKC1遺伝子の変異によって引き起こされる。シュードウリジンは、ヒト免疫不全ウイルス(HIV)の感染におけるウイルスの潜伏過程の調節因子として認識されている[14]。シュードウリジル化は母系遺伝するミトコンドリア糖尿病(MIDD)の発症とも関係している。ミトコンドリアのtRNAの点変異によってあるヌクレオチドのシュードウリジル化が防がれ、tRNAの三次構造が変化しているようである。これによってtRNAの不安定性が高まり、ミトコンドリアでの翻訳と呼吸の欠乏が引き起こされる[14]。
出典
編集- ^ IUPAC-IUB Commission on Biochemical Nomenclature (1970). “Abbreviations and symbols for nucleic acids, polynucleotides, and their constituents”. Biochemistry 9 (20): 4022–4027. doi:10.1021/bi00822a023. PMC 1179624. PMID 5499957.
- ^ a b c Hamma, Tomoko; Ferré-D'Amaré, Adrian R. (November 2006). “Pseudouridine Synthases”. Chemistry & Biology 13 (11): 1125–1135. doi:10.1016/j.chembiol.2006.09.009. ISSN 1074-5521. PMID 17113994.
- ^ a b c d e f g Gray, Michael Charette, Michael W. (2000-05-01). “Pseudouridine in RNA: What, Where, How, and Why”. IUBMB Life 49 (5): 341–351. doi:10.1080/152165400410182. ISSN 1521-6543. PMID 10902565.
- ^ “The spliced leader-associated RNA is a trypanosome-specific sn(o) RNA that has the potential to guide pseudouridine formation on the SL RNA”. RNA 8 (2): 237–246. (2002). doi:10.1017/S1355838202018290. PMC 1370245. PMID 11911368.
- ^ Ge, Junhui; Yu, Yi-Tao (April 2013). “RNA pseudouridylation: new insights into an old modification”. Trends in Biochemical Sciences 38 (4): 210–218. doi:10.1016/j.tibs.2013.01.002. ISSN 0968-0004. PMC 3608706. PMID 23391857.
- ^ a b c d e f g h i j k l Rintala-Dempsey, Anne C.; Kothe, Ute (2017-01-03). “Eukaryotic stand-alone pseudouridine synthases – RNA modifying enzymes and emerging regulators of gene expression?”. RNA Biology 14 (9): 1185–1196. doi:10.1080/15476286.2016.1276150. ISSN 1547-6286. PMC 5699540. PMID 28045575.
- ^ Wu, Guowei; Radwan, Mohamed K.; Xiao, Mu; Adachi, Hironori; Fan, Jason; Yu, Yi-Tao (2016-06-07). “TheTORsignaling pathway regulates starvation-induced pseudouridylation of yeast U2 snRNA”. RNA 22 (8): 1146–1152. doi:10.1261/rna.056796.116. ISSN 1355-8382. PMC 4931107. PMID 27268497.
- ^ Adachi, Hironori; De Zoysa, Meemanage D.; Yu, Yi-Tao (March 2019). “Post-transcriptional pseudouridylation in mRNA as well as in some major types of noncoding RNAs”. Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms 1862 (3): 230–239. doi:10.1016/j.bbagrm.2018.11.002. ISSN 1874-9399. PMC 6401265. PMID 30414851.
- ^ a b Penzo, M.; Guerrieri, A. N.; Zacchini, F.; Treré, D.; Montanaro, L. (2017-11-01). “RNA Pseudouridylation in Physiology and Medicine: For Better and for Worse”. Genes 8 (11): 301. doi:10.3390/genes8110301. ISSN 2073-4425. PMC 5704214. PMID 29104216.
- ^ Keffer-Wilkes, Laura Carole; Veerareddygari, Govardhan Reddy; Kothe, Ute (2016-11-14). “RNA modification enzyme TruB is a tRNA chaperone”. Proceedings of the National Academy of Sciences 113 (50): 14306–14311. doi:10.1073/pnas.1607512113. ISSN 0027-8424. PMC 5167154. PMID 27849601.
- ^ Xu, J.; Gu, A. Y.; Thumati, N. R.; Wong JMY (2017-09-05). “Quantification of Pseudouridine Levels in Cellular RNA Pools with a Modified HPLC-UV Assay”. Genes 8 (9): 219. doi:10.3390/genes8090219. ISSN 2073-4425. PMC 5615352. PMID 28872587.
- ^ Durairaj, Anita; Limbach, Patrick A. (2008-04-07). “Improving CMC-derivatization of pseudouridine in RNA for mass spectrometric detection”. Analytica Chimica Acta 612 (2): 173–181. doi:10.1016/j.aca.2008.02.026. ISSN 1873-4324. PMC 2424252. PMID 18358863.
- ^ Kalsotra, Auinash (2016-11-02). Faculty of 1000 evaluation for Transcriptome-wide mapping reveals widespread dynamic-regulated pseudouridylation of ncRNA and mRNA.. doi:10.3410/f.718875945.793524920.
- ^ a b Zhao, Yang; Karijolich, John; Glaunsinger, Britt; Zhou, Qiang (October 2016). “Pseudouridylation of 7 SK sn RNA promotes 7 SK sn RNP formation to suppress HIV ‐1 transcription and escape from latency”. EMBO Reports 17 (10): 1441–1451. doi:10.15252/embr.201642682. ISSN 1469-221X. PMC 5048380. PMID 27558685.