U2 snRNA
U2 snRNA(U2 small nuclear RNA)は、事実上すべての真核生物のメジャースプライソソーム(Sm)に存在するsnRNAの一種である。In vivoでは、U2 snRNAは関連ポリペプチドとともに組み立てられてU2 snRNPを形成する。U2 snRNPはメジャースプライソソーム複合体の必須の構成要素である[1]。メジャースプライソソームによるスプライシング経路はU2依存的経路と呼ばれることもあるが、それは一次転写産物に存在するSmイントロンはスプライソソームの組み立ての初期段階において、専らU2 snRNPによって認識されるためである[2]。イントロンの認識に加えて、U2 snRNAはpre-RNAのスプライシング反応の触媒にも関与すると考えられている[3][4]。リボソームRNAと同様に、SmクラスのsnRNAはRNA-RNA間相互作用とRNA-タンパク質間相互作用の双方を媒介しなければならず、これらのタイプの相互作用を促進するため特殊化し高度に保存された一次・二次構造エレメントを進化させている[5][6]。
| U2 snRNA | |
|---|---|
 | |
| 識別 | |
| 略称 | U2 |
| Rfam | RF00004 |
| その他のデータ | |
| リボ核酸の種類 | 遺伝子; snRNA; RNAスプライシング |
| ドメイン | 真核生物 |
| GO | 0000370 0045131 0005686 |
| SO | 0000392 |
| PDB構造 | PDBe |
mRNA一次転写産物のタンパク質コード領域がタンパク質をコードしない長い配列(イントロン)によって分割されていることがフィリップ・シャープとリチャード・ロバーツによって発見された[7][8]直後、ジョーン・A・スタイツはイントロン除去の生化学的機構の特徴づけに取り掛かった[9]。興味深いことに、U1 snRNAの5'領域に見られる配列がhnRNA転写産物の5'スプライスジャンクションの保存配列と広範囲にわたって塩基対の相補性を示すことが観察され、ある種のsnRNAがRNA-RNA間相互作用を介してスプライス部位の境界の認識に関与している可能性が推測された。これらの相互作用の複雑さは当時はまだ十分に理解されていなかったが、近年になって原子分解能の結晶構造が明らかとなり、当初の予想が実際に正しかったことが実証された[5][6][10]。
U2 snRNA認識エレメント
編集出芽酵母Saccharomyces cerevisiaeでは、U2 snRNAは18個のポリペプチドと結合しており、そのうち7つは全てのSmクラスのsnRNPに共通の構造タンパク質である[11]。これらの非特異的構造タンパク質は、RNA内に位置するSm結合部位と呼ばれる高度に保存された認識配列(AUnG、n=4–6) を介してSmクラスのsnRNAと結合する[12]。他の2つのタンパク質A'とB''はU2特異的であり、snRNPの組み立てのためにU2 snRNAに特有の構造エレメント(具体的には3'側の2つのステムループ)を必要とする[11]。3つのサブユニットからなるSF3a複合体、6つのサブユニットからなるSF3b複合体もU2 snRNAと結合する[13]。
U2 snRNAは、一次転写産物の3'スプライス部位の18–40ヌクレオチド上流に位置する、分枝点配列(branch point sequence、BPS)として知られる7–12ヌクレオチドの配列を介して、イントロンの認識に関与している。酵母では、BPSのコンセンサス配列の長さは7ヌクレオチドであり、U2 snRNA側の相補的認識配列は6ヌクレオチドである。これら2つの配列が二重らせんを形成することで、保存されたBPSの5位のアデノシン残基はバルジを形成する。バルジを形成したアデノシン残基はC3'-endo型の立体配座をとり[14]、スプライシング因子Cwc25、Yju2、Isy1の助けのもと、5'スプライス部位のリン原子への求核攻撃(in-line攻撃)のために2'-OHを整列させる[15]。求核攻撃は2段階のエステル交換反応の1段階目を開始し、一般的でない2'-5'-3'連結型のラリアット中間体としてイントロンを切り出す。2段階目のエステル交換反応は2つの隣接するエクソンのライゲーションを伴うものである。
一次構造と二次構造
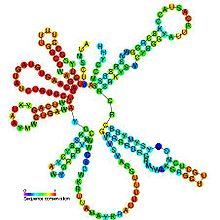
編集U2 snRNAの配列の長さには真核生物種の間で最大1桁程度の差異が存在するが、すべてのU2 snRNA、特に5'末端の最初の80ヌクレオチドには系統学的に不変の領域が多く含まれており、そこでは85%の部位で配列が保存されている[16]。さらに、ステムループI、II、III、IVを含むいくつかの二次構造エレメントや、これらのドメインを連結する一本鎖領域の一部も保存されている[16][17]。酵母のU2 snRNAのステムループIIには一般的でないGA塩基対が含まれており、特徴的なUターンループモチーフが形成される。このモチーフはtRNAのアンチコドンループに類似した幾何学的コンフォメーションをとる[5]。すべてのU2 snRNAにはターミナルステムループ(ステムループIV)が存在する。このステムループは10–16塩基対のヘリックスと11ヌクレオチドのループからなり、ループ部分のコンセンサス配列は5'-UYGCANUURYN-3'である[16]。
U2 snRNAは全てのsnRNAのなかで最も広範囲に修飾を受けている[18]。こうした転写後修飾の正確な位置は生物種によって異なるが、U2 snRNAの修飾と生物学的機能には強い相関が存在することが示唆されている[18]。修飾には、一部のウリジン残基のシュードウリジンへの変換、2'-O-メチル化、核酸塩基のメチル化、5'モノメチル化グアノシンキャップの2,2,7-トリメチル化グアノシンキャップへの変換が含まれる[18]。こうした修飾の多くは分子の5'末端の27ヌクレオチドの領域内にみられる[18]。
コンフォメーションのダイナミクス
編集スプライソソームは、その組み立てとスプライシング過程を通じてコンフォメーションの再編成が何度か起こる、動的な分子機械である。スプライソソームの再編成に関する生化学的詳細の多くはいまだ不明であるが、近年の研究により、スプライシング反応の第一段階を進行させる、重要なU2 snRNAとU6 snRNAのフォールディング複合体の形成が可視化されている[6][19]。このフォールディングは4ヘリックスジャンクションの形成を促進し、活性部位の重要な構成要素のための足場を提供すると考えられており、5'スプライス部位への分枝点アデノシンの2'OHによる求核攻撃のための整列と、負電荷の形成を安定化するための2つのMg2+イオンの配位のために利用される[19]。
進化的起源
編集U2とU6の間のフォールディングの特筆すべき特徴は、自己スプライシンググループIIイントロンのドメインVとの構造的類似性である[3][6]。U6 snRNAに存在するAGCトライアドはグループIIイントロンでも保存されており、同様に三次元的スタッキングを促進することが知られている[6]。U2-U6フォールディングの初期におけるGUゆらぎ塩基対の形成は、グループIIイントロンの触媒コアの形成時にも観察されている[19]。最後に、U2-U6フォールド内での金属結合部位の構造的保存性を考えると、スプライソソームはグループIIイントロンと同じ2金属イオン機構を利用している可能性が高い[3]。こうしたグループIIイントロンとスプライソソームの活性部位のU2-U6フォールドとの二次・三次構造的保存性は、グループIIイントロンとスプライソソームが共通の進化的起源を持つことを示唆している。
出典
編集- ^ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2002). Molecular Biology of the Cell (4th ed.). Garland Science. ISBN 978-0815332183
- ^ Nelson, David L; Cox, Michael M; Lehninger, Albert L (2013). Lehninger principles of biochemistry (6th ed.). New York: W.H. Freeman and Company. ISBN 9781429234146. OCLC 824794893
- ^ a b c “RNA catalyses nuclear pre-mRNA splicing”. Nature 503 (7475): 229–34. (November 2013). Bibcode: 2013Natur.503..229F. doi:10.1038/nature12734. PMC 4666680. PMID 24196718.
- ^ “The Spliceosome: A Protein-Directed Metalloribozyme”. Journal of Molecular Biology 429 (17): 2640–2653. (August 2017). doi:10.1016/j.jmb.2017.07.010. PMID 28733144.
- ^ a b c Stallings, Sarah C; Moore, Peter B (1997). “The structure of an essential splicing element: stem loop IIa from yeast U2 snRNA”. Structure 5 (9): 1173–1185. doi:10.1016/s0969-2126(97)00268-2. ISSN 0969-2126. PMID 9331416.
- ^ a b c d e “Structure of the yeast U2/U6 snRNA complex”. RNA 18 (4): 673–83. (April 2012). doi:10.1261/rna.031138.111. PMC 3312555. PMID 22328579.
- ^ “Spliced segments at the 5' terminus of adenovirus 2 late mRNA”. Proceedings of the National Academy of Sciences of the United States of America 74 (8): 3171–5. (August 1977). Bibcode: 1977PNAS...74.3171B. doi:10.1073/pnas.74.8.3171. PMC 431482. PMID 269380.
- ^ “An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA”. Cell 12 (1): 1–8. (September 1977). doi:10.1016/0092-8674(77)90180-5. PMID 902310.
- ^ “Are snRNPs involved in splicing?”. Nature 283 (5743): 220–4. (January 1980). doi:10.1038/283220a0. PMID 7350545.
- ^ “Invariant U2 snRNA nucleotides form a stem loop to recognize the intron early in splicing”. Molecular Cell 38 (3): 416–27. (May 2010). doi:10.1016/j.molcel.2010.02.036. PMC 2872779. PMID 20471947.
- ^ a b “U2 snRNA sequences that bind U2-specific proteins are dispensable for the function of U2 snRNP in splicing”. Genes & Development 3 (12A): 1887–98. (December 1989). doi:10.1101/gad.3.12a.1887. PMID 2559872.
- ^ “Monospecific antibodies reveal details of U2 snRNP structure and interaction between U1 and U2 snRNPs”. The EMBO Journal 5 (5): 997–1002. (May 1986). doi:10.1002/j.1460-2075.1986.tb04314.x. PMC 1166893. PMID 2941274.
- ^ Dziembowski, Andrzej; Ventura, Ana-Paula; Rutz, Berthold; Caspary, Friederike; Faux, Céline; Halgand, Frédéric; Laprévote, Olivier; Séraphin, Bertrand (2004-12-08). “Proteomic analysis identifies a new complex required for nuclear pre-mRNA retention and splicing”. The EMBO Journal 23 (24): 4847–4856. doi:10.1038/sj.emboj.7600482. ISSN 0261-4189. PMC 535094. PMID 15565172.
- ^ “Crystal structure of a model branchpoint-U2 snRNA duplex containing bulged adenosines”. RNA 7 (5): 682–91. (May 2001). doi:10.1017/S1355838201002187. PMC 1370120. PMID 11350032.
- ^ Galej, Wojciech P.; Wilkinson, Max E.; Fica, Sebastian M.; Oubridge, Chris; Newman, Andrew J.; Nagai, Kiyoshi (8 September 2016). “Cryo-EM structure of the spliceosome immediately after branching”. Nature 537 (7619): 197–201. Bibcode: 2016Natur.537..197G. doi:10.1038/nature19316. ISSN 1476-4687. PMC 5156311. PMID 27459055.
- ^ a b c “Spliceosomal snRNAs”. Annual Review of Genetics 22: 387–419. (1988). doi:10.1146/annurev.ge.22.120188.002131. PMID 2977088.
- ^ “Analysis of RNase-A-resistant regions of adenovirus 2 major late precursor-mRNA in splicing extracts reveals an ordered interaction of nuclear components with the substrate RNA”. Journal of Molecular Biology 196 (3): 559–73. (August 1987). doi:10.1016/0022-2836(87)90032-5. PMID 3681967.
- ^ a b c d “Modifications of U2 snRNA are required for snRNP assembly and pre-mRNA splicing”. The EMBO Journal 17 (19): 5783–95. (October 1998). doi:10.1093/emboj/17.19.5783. PMC 1170906. PMID 9755178.
- ^ a b c “U2-U6 RNA folding reveals a group II intron-like domain and a four-helix junction”. Nature Structural & Molecular Biology 11 (12): 1237–42. (December 2004). doi:10.1038/nsmb863. PMID 15543154.
関連文献
編集- “A conserved pseudouridine modification in eukaryotic U2 snRNA induces a change in branch-site architecture”. RNA 7 (6): 833–45. (June 2001). doi:10.1017/S1355838201002308. PMC 1370140. PMID 11424937.
- “Crystal structure of a model branchpoint-U2 snRNA duplex containing bulged adenosines”. RNA 7 (5): 682–91. (May 2001). doi:10.1017/S1355838201002187. PMC 1370120. PMID 11350032.