アルファプロテオバクテリア綱
アルファプロテオバクテリア綱(アルファプロテオバクテリアこう、Alphaproteobacteria)は細菌ドメインのPseudomonadota門の綱の一つである[19]。生態は光合成細菌から植物共生菌まで非常に多様である。ミトコンドリアの祖先にあたる生物が属していたグループとしても知られ、アルファプロテオバクテリア綱の進化についての研究は真核生物の起源を探る上で重要である。グラム陰性であるが、細胞内寄生種はペプチドグリカンを持たず、したがってグラム染色試験での結果は陽性になることもある[20]。ベータプロテオバクテリアおよびガンマプロテオバクテリアと近縁である。
| アルファプロテオバクテリア綱 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
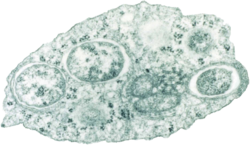
 タイプ属カウロバクター属 (Caulobacter) のタイプ種カウロバクター・ビブリオイデス (Caulobacter vibrioides) の顕微鏡画像。鞭毛を持つ遊走細胞と、付属器を伸ばす有柄細胞とが結合している。
| |||||||||
| 分類 | |||||||||
| |||||||||
| 学名 | |||||||||
| Alphaproteobacteria Garrity et al. 2006[1] (IJSEMリストに記載 2006)[2] | |||||||||
| タイプ属 | |||||||||
| カウロバクター属 Caulobacter Henrici and Johnson 1935[3] (IJSEMリストに記載 1980)[4] 修正 Abraham et al. 1999[5] (IJSEMリストに記載 1980)[6] | |||||||||
| シノニム | |||||||||
| 下位分類(目) | |||||||||
|
本文を参照 |
特徴
編集アルファプロテオバクテリア綱は非常に多様な生物群である。ある属は光栄養細菌であり、その一部はC1化合物を代謝する(例: Methylobacterium spp.)。またある属は植物共生細菌であり(例: Rhizobium spp.)、ある属は節足動物の内生菌であり(Wolbachia属)、ある属は細胞内侵襲性の感染菌である(例: Rickettsia属)。すでに絶滅したが、アルファプロテオバクテリア綱の細菌にはかつて真核生物の細胞に侵入して現在のミトコンドリアとなったプロトミトコンドリアがいた(細胞内共生説)[21]。Rhizobium radiobacter(過去の Agrobacterium tumefaciens)は植物細胞に外来DNAを転移させるために用いられる[22]。Pelagibacter ubiqueといった好気性の非酸素光栄養細菌はアルファプロテオバクテリア綱であり、外洋の微生物コミュニティの10 %以上を構成すると考えられている海中プランクトンである。
下位分類(目)
編集アルファプロテオバクテリア綱の系統関係については大まかな合意はあるものの、いくつかの目 (order) については議論が続いている(例えばペラジバクター目、 リケッチア目、ホロスポラ目)[21]。他の細菌や古細菌と同様、その分類や系統関係については現在も絶えず改定が続いている[23][24]。NCBIのデータベースでは現在20の目が設定されている[25]。一方、GTDBでは35の目が設定されている[26]。分子系統解析とアルファプロテオバクテリア綱メンバーのゲノムに特徴的なインデルの分析[27]により、アルファプロテオバクテリア綱の分岐は細菌ドメインの中ではかなり遅く、ベータプロテオバクテリア綱とガンマプロテオバクテリア綱のみがアルファプロテオバクテリア綱より後に分岐したと推定されている[28][29]。
アルファプロテオバクテリア綱のうち、最も早くに分岐したのはマグネトコッカス目 (Magnetococcales) であることはおおむね支持されているが、この目をアルファプロテオバクテリア綱に含めるかについては議論があり、独立した目としてイータプロテオバクテリア綱 (Etaproteobacteria) の設立も提案されている[30][31](ただし、2021年現在も認められていない)。2018年の研究では、ミトコンドリアはマグネトコッカス目を除く全てのアルファプロテオバクテリア綱に対して姉妹群となることが示唆された[32]。これはミトコンドリアにつながる祖先の系統分岐がアルファプロテオバクテリア綱内で非常に早く起こったことを示している。またミトコンドリアの系統が、リケッチア目と近縁ではあるものの、独立した系統群を形成することも示唆している。
以下に2024年9月現在での所属目について記載する[20]。
IJSEMに承認された目
編集- Caulobacterales カウロバクター目
- Henrici & Johnson 1935[3](IJSEMリストに掲載 1980)[4]
- タイプ属はCaulobacter属(カウロバクター属)である。
- Caulobacteraceae科(カウロバクター科)
- Caulobacter属、Asticcacaulis属(アスティカカウリス属)、Brevundimonas(ブレブンディモナス属)、Phenylobacterium(フェニロバクテリウム属)などを含む。
- Hyphomonadaceae科(ヒフォモナス科)
- Maricaulaceae科(マリカウリス科)
- Caulobacteraceae科(カウロバクター科)
- Emcibacterales
- Futianiales
- Holosporales ホロスポラ目
- Hyphomicrobiales ヒフォミクロビウム目
- Douglas 1957[38](IJSEMリストに掲載 1980)[4]、修正 Hördt et al. 2020[23]
- タイプ属はHyphomicrobium属(ヒフォミクロビウム属)である。
- Hyphomicrobiaceae科(ヒフォミクロビウム科)
- Hyphomicrobium属、Filomicrobium属(フィロミクロビウム属)、Dichotomicrobium属(ジコトミクロビウム属)、Seliberia属(セリベリア属)などを含む。
- Nitrobacteraceae科(ニトロバクター科)
- Nitrobacter属(ニトロバクター属)、Bradyrhizobium属(ブラディリゾビウム属)、Rhodopseudomonas属(ロドシュードモナス属)などを含む。
- Rhizobiaceae科(リゾビウム科)
- Rhizobium属(リゾビウム属)、Agrobacterium(アグロバクテリウム属)、Allorhizobium(アロリゾビウム属)などを含む。
- Kaistiaceae科(カイステア科)
- Kaistia属(カイステア属)とBauldia属で構成される。
- Xanthobacteraceae科(キサントバクター科)
- Hyphomicrobiaceae科(ヒフォミクロビウム科)
- その他、多数の科で構成される。
- Iodidimonadales
- Kordiimonadales コーディイモナス目
- Magnetococcales マグネトコッカス目
- Mariprofundales マリプロフンドゥス目
- Hördt et al. 2020[23](IJSEMリストに掲載 2020)[41]
- Mariprofundus属(マリプロフンドゥス属)のみを含むMariprofundaceae科(マリプロフンドゥス科)のみで構成される。
- Micropepsales
- Minwuiales
- Rhodobacterales ロドバクター目
- Garrity et al. 2006[48](IJSEMリストに記載 2006)[2]、修正 Hördt et al. 2020[23](IJSEMリストに掲載 2020)[41]
- タイプ属はRhodobacter属(ロドバクター属)である。
- Paracoccaceae科(パラコッカス科)
- Paracoccus属(パラコッカス属)、Rhodobacter属、Pseudorhodobacter属(シュードロドバクター属)などを含む。
- Neomegalonemataceae科
- Neomegalonema属のみを含む。
- Roseobacteraceae科(ロゼオバクター科)
- Roseobacter属(ロゼオバクター属)、Antarctobacter属(アンタルクトバクター属)、Octadecabacter属(オクタデカバクター属)、Roseovarius属(ロゼオバリウス属)、Roseivivax属(ロゼイビバックス属)、Sagittula属(サギッツラ属)、Ruegeria属(ルエゲリア属)などを含む。
- Paracoccaceae科(パラコッカス科)
- その他、多数の科で構成される。
- Rhodospirillales ロドスピリルム目
- Pfennig and Trüper 1971[49](IJSEMリストに掲載 1980)[4]、修正 Hördt et al. 2020[23](IJSEMリストに掲載 2020)[41]
- タイプ属はRhodospirillum属(ロドスピリルム属)である。
- Rhodospirillaceae科(ロドスピリルム科)
- Rhodospirillum属、Magnetospirillum属(マグネトスピリルム属)、Rhodospira属(ロドスピラ属)、Roseospira属(ロセオスピラ属)などを含む。
- Acetobacteraceae科(アセトバクター科)
- Acetobacter属(アセトバクター属)、Gluconacetobacter属(グルコナセトバクター属)、Gluconobacter属(グルコノバクター属)、Acidomonas属(アシドモナス属)などを含む。
- Rhodospirillaceae科(ロドスピリルム科)
- その他、多数の科で構成される。
- Rhodothalassiales ロドタラシウム目
- Rickettsiales リケッチア目
- Sneathiellales スネアチエラ目
- Sphingomonadales スフィンゴモナス目
- Yabuuchi and Kosako 2006[59](IJSEMリストに掲載 2006)[2]、修正 Hördt et al. 2020[23] (IJSEMリストに掲載 2020)[41]
- タイプ属はSphingomonas属(スフィンゴモナス属)である。
- Sphingomonadaceae科(スフィンゴモナス科)
- Sphingobium属、Sphingopyxis属(スフィンゴピクシス属)、Blastomonas属(ブラストモナス属)などを含む。
- Sphingosinicellaceae科(スフィンゴシニセラ科)
- Sphingosinicella属(スフィンゴシニセラ属)などを含む。
- Erythrobacteraceae科(エリスロバクター科)
- Erythrobacter属(エリスロバクター属)、Novosphingobium属(ノボスフィンゴビウム属)などを含む。
- Zymomonadaceae科(ザイモモナス科)
- Zymomonas属(ザイモモナス属)のみで構成される。
- Sphingomonadaceae科(スフィンゴモナス科)
IJSEMに未承認の目
編集- “Parvularculales” パルブラルキュラ目
- “Candidatus Pelagibacterales” ペラギバクター目
- “Candidatus Puniceispirillales”
- Chuvochina et al. 2023[16]
- “Candidatus Puniceispirillum” 属のみを含む “Candidatus Puniceispirillaceae” 科のみで構成される。
系統樹
編集アルファプロテオバクテリア綱の分類体系は “List of Prokaryotic names with Standing in Nomenclature” (LPSN)[20]と米国立生物工学情報センター (NCBI)[25]で公開されている。下記の系統樹は呼吸複合体IのサブユニットNuoLを用いて2022年に作成されたものである[65]。
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
自然遺伝子形質転換
編集アルファプロテオバクテリア綱の自然遺伝子形質転換の過程についてはAgrobacterium tumefaciens[66]、Methylobacterium organophilum[67]、およびBradyrhizobium japonicum[68]で明らかになっている。形質転換はDNAをある細胞から別の細胞に移動させる過程であり、相同組換えによって供給DNAは受容ゲノムに組み込まれる。
脚注
編集- ^ Garrity GM, Bell JA, Lilburn T (2005). “Class I. Alphaproteobacteria class. nov.”. In Brenner DJ, Krieg NR, Staley JT, Garrity GM. Bergey's Manual of Systematic Bacteriology, 2nd edn, vol. 2 (The Proteobacteria), part C (The Alpha-, Beta-, Delta-, and Epsilonproteobacteria). Springer (New York). p. 1
- ^ a b c “List of new names and new combinations previously effectively, but not validly, published”. International Journal of Systematic and Evolutionary Microbiology 56 (1): 1-6. (01 January 2006). doi:10.1099/ijs.0.64188-0.
- ^ a b Arthur T. Henrici, Delia E. Johnson (1 July 1935). “Studies of Freshwater Bacteria: II. Stalked Bacteria, a New Order of Schizomycetes”. Journal of Bacteriology 30 (1): 61-93. doi:10.1128/jb.30.1.61-93.1935. PMC 543637. PMID 16559821.
- ^ a b c d e V. B. D. Skerman, Vicki. McGOWAN, P. H. A. Sneath (01 January 1980). “Approved Lists of Bacterial Names”. International Journal of Systematic and Evolutionary Microbiology 30 (1): 225-420. doi:10.1099/00207713-30-1-225.
- ^ Wolf-Rainer Abraham, Carsten Strömpl, Holger Meyer, Sabine Lindholst, Edward R. B. Moore, Ruprecht Christ, Marc Vancanneyt, B. J. Tindall, Antonio Bennasar, John Smit and Michael Tesar (01 July 1999). “Phylogeny and polyphasic taxonomy of Caulobacter species. Proposal of Maricaulis gen. nov. with Maricaulis maris (Poindexter) comb. nov. as the type species, and emended description of the genera Brevundimonas and Caulobacter”. International Journal of Systematic and Evolutionary Microbiology 49 (3): 1053-73. doi:10.1099/00207713-49-3-1053. PMID 10425763.
- ^ Anonymous (01 October 1999). “Notification that new names and new combinations have appeared in volume 49, part 3, of the IJSB”. International Journal of Systematic and Evolutionary Microbiology 49 (4): 1327-1328. doi:10.1099/00207713-49-4-1327.
- ^ T Cavalier-Smith (01 January 2002). “The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification”. International Journal of Systematic and Evolutionary Microbiology 52 (1): 7-76. doi:10.1099/00207713-52-1-7. PMID 11837318.
- ^ “Notification list. Notification that new names and new combinations have appeared in volume 52, part 1 of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 52: 293-295. (2002). doi:10.1099/ijs.0.02318-0.
- ^ N. E. GIBBONS and R. G. E. MURRAY (01 January 1978). “Proposals Concerning the Higher Taxa of Bacteria”. International Journal of Systematic and Evolutionary Microbiology 28 (1): 1-6. doi:10.1099/00207713-28-1-1.
- ^ Murray RGE (1984). “The higher taxa, or, a place for everything..?”. In Krieg NR, Holt JG. Bergey's manual of systematic bacteriology, vol. 1.. The Williams & Wilkins Co., Baltimore. pp. 31-34
- ^ “Validation of the Publication of New Names and New Combinations Previously Effectively Published Outside the IJSB”. International Journal of Systematic and Evolutionary Microbiology 38 (2): 220-222. (01 April 1988). doi:10.1099/00207713-38-2-220.
- ^ a b David Emerson, Jeremy A Rentz, Timothy G Lilburn, Richard E Davis, Henry Aldrich, Clara Chan, Craig L Moyer (2007 Aug 1). “A novel lineage of proteobacteria involved in formation of marine Fe-oxidizing microbial mat communities”. PLoS One 2 (7): e667. doi:10.1371/journal.pone.0000667. PMC 1930151. PMID 17668050.
- ^ a b Aharon Oren, George M. Garrity, Charles T. Parker, Maria Chuvochina and Martha E. Trujillo (30 June 2020). “Lists of names of prokaryotic Candidatus taxa”. International Journal of Systematic and Evolutionary Microbiology 70 (7): 3956-4042. doi:10.1099/ijsem.0.003789.
- ^ Aharon Oren (01 April 2017). “A plea for linguistic accuracy – also for Candidatus taxa”. International Journal of Systematic and Evolutionary Microbiology 67 (4). doi:10.1099/ijsem.0.001715.
- ^ a b Thomas Cavalier-Smith, Ema E-Yung Chao (03 January 2020). “Multidomain ribosomal protein trees and the planctobacterial origin of neomura (eukaryotes, archaebacteria)”. Protoplasma 257 (3): 621–753. doi:10.1007/s00709-019-01442-7. PMC 7203096. PMID 31900730.
- ^ a b Maria Chuvochina, Aaron J Mussig, Pierre-Alain Chaumeil, Adam Skarshewski, Christian Rinke, Donovan H Parks, Philip Hugenholtz (21 July 2023). “Proposal of names for 329 higher rank taxa defined in the Genome Taxonomy Database under two prokaryotic codes”. FEMS Microbiology Letters 370: fnad071. doi:10.1093/femsle/fnad071. PMC 10408702. PMID 37480240.
- ^ a b Aharon Oren and Markus Göker (01 February 2024). “Validation List no. 215. Valid publication of new names and new combinations effectively published outside the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 74 (1): 6173. doi:10.1099/ijsem.0.006173.
- ^ Hiroko Makita, Emiko Tanaka, Satoshi Mitsunobu, Masayuki Miyazaki, Takuro Nunoura, Katsuyuki Uematsu, Yoshihiro Takaki, Shinro Nishi, Shigeru Shimamura & Ken Takai (20 October 2016). “Mariprofundus micogutta sp. nov., a novel iron-oxidizing zetaproteobacterium isolated from a deep-sea hydrothermal field at the Bayonnaise knoll of the Izu-Ogasawara arc, and a description of Mariprofundales ord. nov. and Zetaproteobacteria classis nov.”. Archives of Microbiology 199: 335–346. doi:10.1007/s00203-016-1307-4. PMID 27766355.
- ^ Brenner, Don J.; Krieg, Noel R.; Staley, James T. (July 26, 2005) [1984(Williams & Wilkins)]. George M. Garrity. ed. The Proteobacteria. Bergey's Manual of Systematic Bacteriology. 2C (2nd ed.). New York: Springer. pp. 1388. ISBN 978-0-387-24145-6. British Library no. GBA561951
- ^ a b c Jean P. Euzéby, Aidan C. Parte. “Class Alphaproteobacteria”. List of Prokaryotic names with Standing in Nomenclature. 2024年9月28日閲覧。
- ^ a b Ferla, Matteo P.; Thrash, J. Cameron; Giovannoni, Stephen J.; Patrick, Wayne M. (2013-12-11). Badger, Jonathan H.. ed. “New rRNA Gene-Based Phylogenies of the Alphaproteobacteria Provide Perspective on Major Groups, Mitochondrial Ancestry and Phylogenetic Instability” (英語). PLoS ONE 8 (12): e83383. doi:10.1371/journal.pone.0083383. ISSN 1932-6203. PMC 3859672. PMID 24349502.
- ^ Chilton MD, Drummond MH, Merio DJ, Sciaky D, Montoya AL, Gordon MP, Nester EW (1977). “Stable incorporation of plasmid DNA into higher plant cells: the molecular basis of crown gall tumorigenesis”. Cell 11 (2): 263–71. doi:10.1016/0092-8674(77)90043-5. PMID 890735.
- ^ a b c d e f g Hördt, Anton; López, Marina García; Meier-Kolthoff, Jan P.; Schleuning, Marcel; Weinhold, Lisa-Maria; Tindall, Brian J.; Gronow, Sabine; Kyrpides, Nikos C. et al. (2020-04-07). “Analysis of 1,000+ Type-Strain Genomes Substantially Improves Taxonomic Classification of Alphaproteobacteria”. Frontiers in Microbiology 11: 468. doi:10.3389/fmicb.2020.00468. ISSN 1664-302X. PMC 7179689. PMID 32373076.
- ^ Muñoz-Gómez, Sergio A; Hess, Sebastian; Burger, Gertraud; Lang, B Franz; Susko, Edward; Slamovits, Claudio H; Roger, Andrew J (2019-02-25). “An updated phylogeny of the Alphaproteobacteria reveals that the parasitic Rickettsiales and Holosporales have independent origins” (英語). eLife 8: e42535. doi:10.7554/eLife.42535. ISSN 2050-084X. PMC 6447387. PMID 30789345.
- ^ a b Sayers. “Alphaproteobacteria”. National Center for Biotechnology Information (NCBI) taxonomy database. 2011年6月5日閲覧。
- ^ “GTDB”. GTDB. 2021年8月27日閲覧。
- ^ Gupta, Radhey S. (2005-01). “Protein Signatures Distinctive of Alpha Proteobacteria and Its Subgroups and a Model for α –Proteobacterial Evolution” (英語). Critical Reviews in Microbiology 31 (2): 101–135. doi:10.1080/10408410590922393. ISSN 1040-841X.
- ^ Gupta R.S. (2000). “Phylogeny of Proteobacteria: Relationships to other eubacterial phyla and to eukaryotes”. FEMS Microbiol. Rev. 24 (4): 367–402. doi:10.1111/j.1574-6976.2000.tb00547.x. PMID 10978543.
- ^ Gupta R.S.; Sneath P.H.A. (2007). “Application of the Character compatibility approach to generalized molecular sequence data: Branching order of the Proteobacterial subdivisions”. J. Mol. Evol. 64 (1): 90–100. doi:10.1007/s00239-006-0082-2. PMID 17160641.
- ^ Ji, Boyang; Zhang, Sheng-Da; Zhang, Wei-Jia; Rouy, Zoe; Alberto, François; Santini, Claire-Lise; Mangenot, Sophie; Gagnot, Séverine et al. (2017-03). “The chimeric nature of the genomes of marine magnetotactic coccoid-ovoid bacteria defines a novel group of P roteobacteria: Genome of marine magnetotactic strain MO-1” (英語). Environmental Microbiology 19 (3): 1103–1119. doi:10.1111/1462-2920.13637.
- ^ Lin, Wei; Zhang, Wensi; Zhao, Xiang; Roberts, Andrew P.; Paterson, Greig A.; Bazylinski, Dennis A.; Pan, Yongxin (2018-06). “Genomic expansion of magnetotactic bacteria reveals an early common origin of magnetotaxis with lineage-specific evolution” (英語). The ISME Journal 12 (6): 1508–1519. doi:10.1038/s41396-018-0098-9. ISSN 1751-7362. PMC 5955933. PMID 29581530.
- ^ Martijn, Joran; Vosseberg, Julian; Guy, Lionel; Offre, Pierre; Ettema, Thijs J. G. (2018-05). “Deep mitochondrial origin outside the sampled alphaproteobacteria” (英語). Nature 557 (7703): 101–105. doi:10.1038/s41586-018-0059-5. ISSN 0028-0836.
- ^ a b Takao Iino, Moriya Ohkuma, Yoichi Kamagata and Seigo Amachi (01 December 2016). “Iodidimonas muriae gen. nov., sp. nov., an aerobic iodide-oxidizing bacterium isolated from brine of a natural gas and iodine recovery facility, and proposals of Iodidimonadaceae fam. nov., Iodidimonadales ord. nov., Emcibacteraceae fam. nov. and Emcibacterales ord. nov.”. International Journal of Systematic and Evolutionary Microbiology 66 (12): 5016-5022. doi:10.1099/ijsem.0.001462. PMID 27566239.
- ^ a b “Notification that new names of prokaryotes, new combinations and new taxonomic opinions have appeared in volume 66, part 12, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 67 (3): 525-528. (01 March 2017). doi:10.1099/ijsem.0.001834.
- ^ Lirui Liu, Wen-Cong Huang, Jie Pan, Jiayi Li, Yuhan Huang, Dayu Zou, Huan Du, Yang Liu, Meng Li (21 December 2022). “Isolation and Genomics of Futiania mangrovii gen. nov., sp. nov., a Rare and Metabolically Versatile Member in the Class Alphaproteobacteria”. ASM Journals 11 (1): e0411022. doi:10.1128/spectrum.04110-22. PMC 9927469. PMID 36541777.
- ^ Franziska Szokoli, Michele Castelli, Elena Sabaneyeva, Martina Schrallhammer, Sascha Krenek, Thomas G. Doak, Thomas U. Berendonk, Giulio Petroni (21 November 2016). “Disentangling the Taxonomy of Rickettsiales and Description of Two Novel Symbionts (“Candidatus Bealeia paramacronuclearis” and “Candidatus Fokinia cryptica”) Sharing the Cytoplasm of the Ciliate Protist Paramecium biaurelia”. ASM Journals 82 (24): 7236-7247. doi:10.1128/AEM.02284-16. PMC 5118934. PMID 27742680.
- ^ Aharon Oren and Markus Göker (25 November 2020). “List of new names and new combinations that have appeared in effective publications outside of the IJSEM and are submitted for valid publication”. International Journal of Systematic and Evolutionary Microbiology 70 (11): 5596-5601. doi:10.1099/ijsem.0.004484.
- ^ Douglas HC (1957). “Order III. Hyphomicrobiales Douglas, Ordo Nov”. In Breed RS, Murray EGD, Smith NR. Bergey’s Manual of Determinative Bacteriology, 7th ed.. The Williams and Wilkins Co., Baltimore. pp. 276-280.
- ^ Kae Kyoung Kwon, Hee-Soon Lee, Sung Hyun Yang and Sang-Jin Kim (01 September 2005). “Kordiimonas gwangyangensis gen. nov., sp. nov., a marine bacterium isolated from marine sediments that forms a distinct phyletic lineage (Kordiimonadales ord. nov.) in the ‘Alphaproteobacteria’”. International Journal of Systematic and Evolutionary Microbiology 55 (5): 2033-2037. doi:10.1099/ijs.0.63684-0. PMID 16166706.
- ^ “Notification that new names and new combinations have appeared in volume 55, part 5, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 56 (1): 7-9. (01 January 2006). doi:10.1099/ijs.0.64166-0.
- ^ a b c d e Aharon Oren, George Garrity (30 July 2020). “Notification of changes in taxonomic opinion previously published outside the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 70 (7): 4061-4090. doi:10.1099/ijsem.0.004245.
- ^ Dennis A. Bazylinski, Timothy J. Williams, Christopher T. Lefèvre, Ryan J. Berg, Chuanlun L. Zhang, Samuel S. Bowser, Annette J. Dean and Terrence J. Beveridge (01 March 2013). “Magnetococcus marinus gen. nov., sp. nov., a marine, magnetotactic bacterium that represents a novel lineage (Magnetococcaceae fam. nov., Magnetococcales ord. nov.) at the base of the Alphaproteobacteria”. International Journal of Systematic and Evolutionary Microbiology 63 (Pt_3): 801-808. doi:10.1099/ijs.0.038927-0. PMID 22581902.
- ^ “Notification that new names and new combinations have appeared in volume 63, part 3, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 63 (Pt_6): 1939-1941. (01 June 2013). doi:10.1099/ijs.0.053207-0.
- ^ Austin B. Harbison, Laiken E. Price, Michael D. Flythe and Suzanna L. Bräuer (01 April 2017). “Micropepsis pineolensis gen. nov., sp. nov., a mildly acidophilic alphaproteobacterium isolated from a poor fen, and proposal of Micropepsaceae fam. nov. within Micropepsales ord. nov.”. International Journal of Systematic and Evolutionary Microbiology 67 (4): 839-844. doi:10.1099/ijsem.0.001681. PMID 27902348.
- ^ Aharon Oren, George Garrity (01 July 2017). “Notification that new names of prokaryotes, new combinations, and new taxonomic opinions have appeared in volume 67, part 4, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 67 (7): 2079-2080. doi:10.1099/ijsem.0.002098.
- ^ Cong Sun, Lin Xu, Xiao-Yun Yu, Zhe Zhao, Yue-Hong Wu, Aharon Oren, Chun-Sheng Wang and Xue-Wei Xu (16 October 2018). “Minwuia thermotolerans gen. nov., sp. nov., a marine bacterium forming a deep branch in the Alphaproteobacteria, and proposal of Minwuiaceae fam. nov. and Minwuiales ord. nov.”. International Journal of Systematic and Evolutionary Microbiology 68 (12): 3856-3862. doi:10.1099/ijsem.0.003073. PMID 30325296.
- ^ Aharon Oren, George Garrity (04 March 2019). “Notification that new names of prokaryotes, new combinations, and new taxonomic opinions have appeared in volume 68, part 12, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 69 (3): 600-601. doi:10.1099/ijsem.0.003197.
- ^ Garrity GM, Bell JA, Lilburn T (2005). “Order III. Rhodobacterales ord. nov.”. In Brenner DJ, Krieg NR, Staley JT, Garrity GM. Bergey's Manual of Systematic Bacteriology, 2nd edn, vol. 2 (The Proteobacteria), part C (The Alpha-, Beta-, Delta-, and Epsilonproteobacteria). Springer, New York. p. 161
- ^ Norbert Pfennig and Hans G. Truper (01 January 1971). “Higher Taxa of the Phototrophic Bacteria”. International Journal of Systematic and Evolutionary Microbiology 21 (1): 17-18. doi:10.1099/00207713-21-1-17.
- ^ V Venkata Ramana, S Kalyana Chakravarthy, E V V Ramaprasad, V Thiel, J F Imhoff, Ch Sasikala, Ch V Ramana (February 2013). “Emended description of the genus Rhodothalassium Imhoff et al., 1998 and proposal of Rhodothalassiaceae fam. nov. and Rhodothalassiales ord. nov”. Systematic and Applied Microbiology 36 (1): 28-32. doi:10.1016/j.syapm.2012.09.003. PMID 23265196.
- ^ Aharon Oren, George Garrity (01 September 2014). “List of new names and new combinations previously effectively, but not validly, published”. International Journal of Systematic and Evolutionary Microbiology 64 (Pt_9): 2927-2929. doi:10.1099/ijs.0.068759-0.
- ^ Gieszczykiewicz M (1939). “Zagadniene systematihki w bakteriologii - Zür Frage der Bakterien-Systematic”. Bulletin de l'Académie Polonaise des Sciences Série des Sciences Biologiques. 1. pp. 9-27
- ^ Don J. Brenner, Steven P. O’Connor, Herbert H. Winkler and Arnold G. Steigerwalt (01 October 1993). “Proposals To Unify the Genera Bartonella and Rochalimaea, with Descriptions of Bartonella quintana comb. nov., Bartonella vinsonii comb. nov., Bartonella henselae comb. nov., and Bartonella elizabethae comb. nov., and To Remove the Family Bartonellaceae from the Order Rickettsiales”. International Journal of Systematic and Evolutionary Microbiology 43 (4): 777-86. doi:10.1099/00207713-43-4-777. PMID 8240958.
- ^ “Notification that New Names and New Combinations Have Appeared in Volume 43, No. 4, of the IJSB”. International Journal of Systematic and Evolutionary Microbiology 44 (1): 184. (01 January 1994). doi:10.1099/00207713-44-1-184.
- ^ J S Dumler, A F Barbet, C P Bekker, G A Dasch, G H Palmer, S C Ray, Y Rikihisa and F R Rurangirwa (01 November 2001). “Reorganization of genera in the families Rickettsiaceae and Anaplasmataceae in the order Rickettsiales: unification of some species of Ehrlichia with Anaplasma, Cowdria with Ehrlichia and Ehrlichia with Neorickettsia, descriptions of six new species combinations and designation of Ehrlichia equi and 'HGE agent' as subjective synonyms of Ehrlichia phagocytophila”. International Journal of Systematic and Evolutionary Microbiology 51 (6): 2145-2165. doi:10.1099/00207713-51-6-2145. PMID 11760958.
- ^ “Notification list. Notification that new names and new combinations have appeared in volume 51, part 6, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 52 (1): 5-6. (01 January 2002). doi:10.1099/00207713-52-1-5.
- ^ Kurahashi M, Fukunaga Y, Harayama S, Yokota A (2008). “Sneathiella glossodoripedis sp. nov., a marine alphaproteobacterium isolated from the nudibranch Glossodoris cincta, and proposal of Sneathiellales ord. nov. and Sneathiellaceae fam. nov.”. Int J Syst Evol Microbiol 58 (Pt 3): 548–552. doi:10.1099/ijs.0.65328-0. PMID 18319453.
- ^ “Notification that new names and new combinations have appeared in volume 58, part 3, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 58 (6): 1273-1274. (01 June 2008). doi:10.1099/ijs.0.2008/003863-0.
- ^ Yabuuchi E, Kosako Y (2005). “Order IV. Sphingomonadales ord. nov.”. In Brenner DJ, Krieg NR, Staley JT, Garrity GM. Bergey's Manual of Systematic Bacteriology, 2nd edn, vol. 2 (The Proteobacteria), part C (The Alpha-, Beta-, Delta-, and Epsilonproteobacteria). Springer, New York. pp. 230-233.
- ^ Jang-Cheon Cho and Stephen J. Giovannoni (01 July 2003). “Parvularcula bermudensis gen. nov., sp. nov., a marine bacterium that forms a deep branch in the α-Proteobacteria”. International Journal of Systematic and Evolutionary Microbiology 53 (4): 1031-1036. doi:10.1099/ijs.0.02566-0. PMID 12892122.
- ^ “Notification that new names and new combinations have appeared in volume 53, part 4, of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 53 (6): 1703-1705. (01 November 2003). doi:10.1099/ijs.0.02972-0.
- ^ Lin-Lin Sun, Yan-Ru Dang, Yi Li, Qi-Long Qin, Hai-Nan Su, Ping-Yi Li, Xiu-Lan Chen, Yu-Zhong Zhang and Xi-Ying Zhang (01 August 2019). “Parvularcula marina sp. nov., isolated from surface water of the South China Sea, and emended description of the genus Parvularcula”. International Journal of Systematic and Evolutionary Microbiology 69 (8): 2571-2576. doi:10.1099/ijsem.0.003543. PMID 31225791.
- ^ Aharon Oren and George M. Garrity (01 November 2019). “Notification that new names of prokaryotes, new combinations, and new taxonomic opinions have appeared in volume 69, part 8 of the IJSEM”. International Journal of Systematic and Evolutionary Microbiology 69 (11): 3315-3317. doi:10.1099/ijsem.0.003692.
- ^ Jana Grote, J. Cameron Thrash, Megan J. Huggett, Zachary C. Landry, Paul Carini, Stephen J. Giovannoni, Michael S. Rappé (18 September 2012). “Streamlining and Core Genome Conservation among Highly Divergent Members of the SAR11 Clade”. ASM Journals 3 (5). doi:10.1128/mbio.00252-12.
- ^ Miguel Cevallos (February 2022). “New Alphaproteobacteria Thrive in the Depths of the Ocean with Oxygen Gradient”. Microorganisms 10 (2): 455. doi:10.3390/microorganisms10020455.
- ^ Demanèche S, Kay E, Gourbière F, Simonet P (2001). “Natural transformation of Pseudomonas fluorescens and Agrobacterium tumefaciens in soil”. Appl. Environ. Microbiol. 67 (6): 2617–21. doi:10.1128/AEM.67.6.2617-2621.2001. PMC 92915. PMID 11375171.
- ^ O'Connor M, Wopat A, Hanson RS (1977). “Genetic transformation in Methylobacterium organophilum”. J. Gen. Microbiol. 98 (1): 265–72. doi:10.1099/00221287-98-1-265. PMID 401866.
- ^ Raina JL, Modi VV (1972). “Deoxyribonucleate binding and transformation in Rhizobium jpaonicum”. Journal of Bacteriology 111 (2): 356–60. doi:10.1128/jb.111.2.356-360.1972. PMC 251290. PMID 4538250.
関連項目
編集外部リンク
編集- ウィキスピーシーズには、アルファプロテオバクテリア綱に関する情報があります。
- Alphaproteobacteria - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
- Bacterial (Prokaryotic) Phylogeny Webpage: Alpha Proteobacteria.